Транскрипция,
ее регуляция,
и как ее изучают
(с помощью секвенирования и биоинформатики)
Ваня Кулаковский, Пущино-Москва
Часть 1: транскрипция у высших эукариот глазами биоинформатика
Что в этом такого и почему это интересно
Assume infinite intelligence and zero prior knowledge.
Attributed to Leo Szilard
Valentine L. Telegdi, "Szilard as Inventor: Accelerators and More", Physics Today 53, 10, 25 (2000); doi: 10.1063/1.1325189...assume that the listener has zero knowledge and infinite intelligence.
Attributed to Max Delbruck
Lily Yeh Jan, "Ion Channels—Molecules in Action", Cell VOLUME 89, ISSUE 6, P829-830, JUNE 13, 1997Тело человека содержит 200-400 (включая почти 150 типов нейронов) типов клеток Harvard BioNumbers ID 103626 and 106155.
Что обеспечивает такое разнообразие?
"Всего лишь" около 19 тысяч белок-кодирующих генов genenames.org, экзоны покрывают лишь 1-2% от полной длины генома (Harvard BioNumbers, PMID 15496913 and 18514361).
Как же
генетическая информация о нескольких десятках тысяч белок-кодирующих генов управляемо реализуется в различных клетках в различных условиях "во времени и пространстве" ?
… связь между геном и внешним признаком заключается не в отдельных независимых цепях реакций, которые ведут от определенного гена к определенному признаку, но значительно сложнее.
… в осуществлении определенного наследственного признака совместно участвуют очень многие, доступные экспериментальному учету отдельные модифицирующие факторы. У нас нет основания и возможности предполагать, что при влиянии на проявление каждого отдельного признака речь идет о специфических, касающихся только данного признака «специальных генах-модификаторах», ибо мы пришли бы к бессмысленному огромному общему числу генов. Таким образом, нужно предположить, что каждый ген в разной степени должен принимать участие в целом ряде процессов развития.
Н.В. Тимофеев-Ресовский
Уровни регуляции
экспрессии генов
Эпигенетика: Упаковка ДНК > Модификации гистонов
> Метилирование ДНК >
Регуляция транскрипции факторами транскрипции
> Альтернативный сплайсинг и эпитранскриптомика > Экспорт, локализация и стабильность РНК > Трансляция белка и пост-трансляционная регуляция
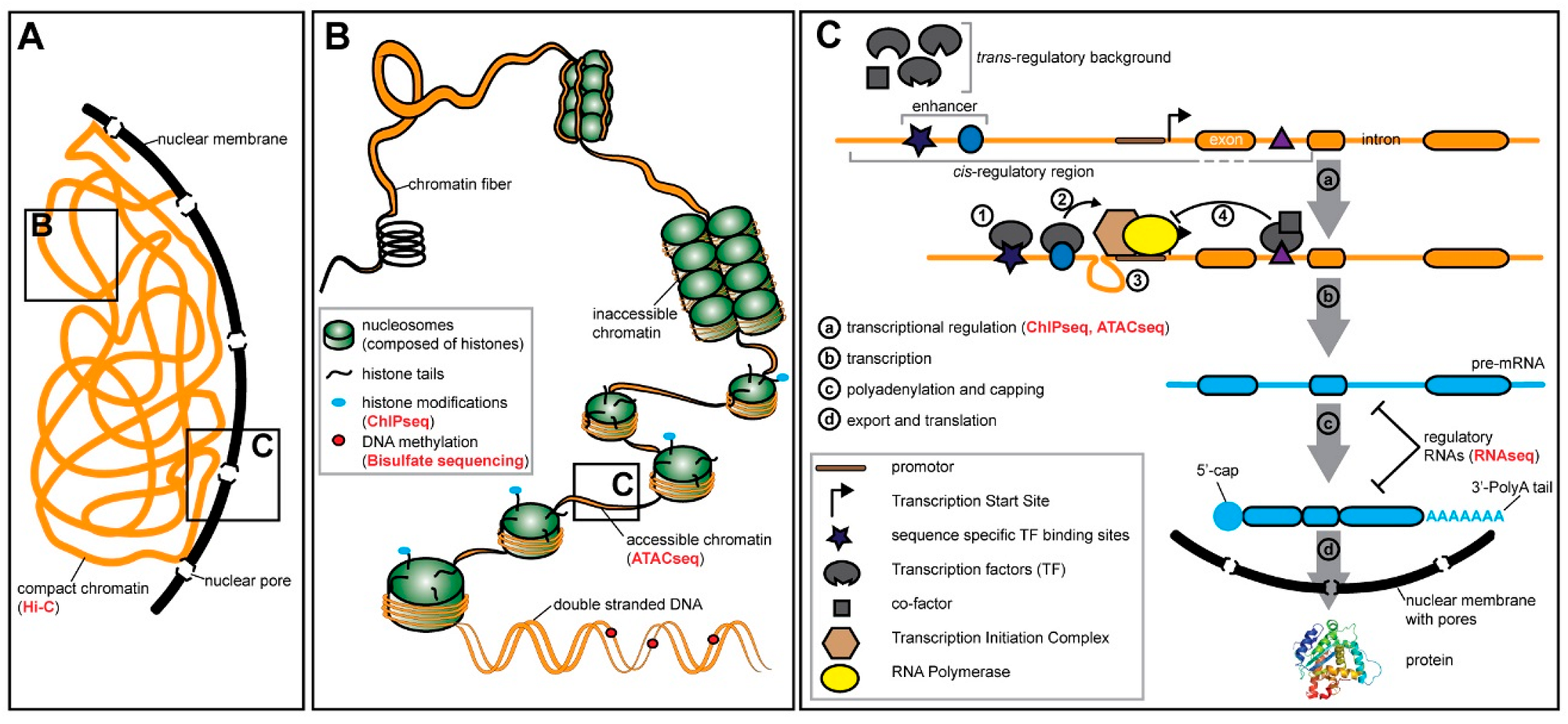 Cloudy with a Chance of Insights: Context Dependent Gene Regulation and Implications for Evolutionary Studies,
Cloudy with a Chance of Insights: Context Dependent Gene Regulation and Implications for Evolutionary Studies, Buchberger et al., Genes, 2019


Нобелевская премия (физиология и медицина) 1968 за "интерпретацию генетического кода и его функции в биосинтезе белка".
Георгий Антонович Гамов

Генетический код
и его полезные свойства:
избыточность и вырожденность > синонимичные и несинонимичные замены > функциональная аннотация мутаций > молекулярные часы > молекулярная эволюция
According to the media sympathetic to science and enthusiastic about sensational discoveries, the “Second Genetic Code” as it was called by New York Times was discovered by Y a-Ming Hou and Paul Schimmel and published in Nature in 1988. It was about recognition of tRNAs by respective aminoacyl-tRNA synthetases. Thirteen years later New Scientist announced the second Second Genetic Code, discovered by Jenuwein and Allis and published in Science. This time it was about histone modifications. Five years later, New York Times, again, reported about “a second code in DNA in addition to the genetic code”. This was already the third Second Genetic Code, discovered by Segal et al., suggesting now nucleosome positioning rules. One, surely, would raise eyebrows having learned that there is also the fourth Second Genetic Code — on interaction specificities between proteins and DNA, and the fifth Second Genetic Code, the name given by Nature magazine to the set of rules governing gene splicing.
Bewildered reader, naturally, would say “I’m done with seconds, can I have a third?”

E.N. Trifonov
Thirty Years of Multiple Sequence Codes, Genomics Proteomics Bioinformatics, 2011Regulated transcription controls the diversity, developmental pathways, and spatial organisation of the hundreds of cell types that make up a mammal.
Проект FANTOM5, посвященный масштабному изучению инициации транскрипции различных типах клеток эукариот
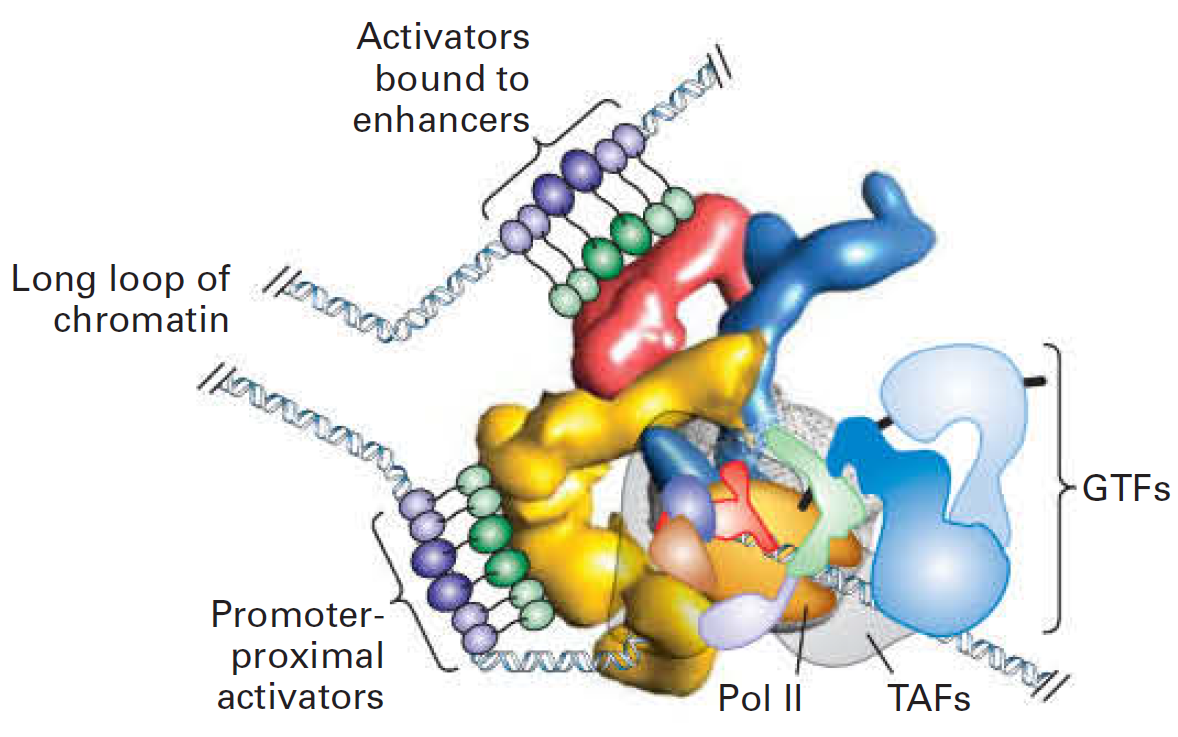
Петлевая модель регуляции

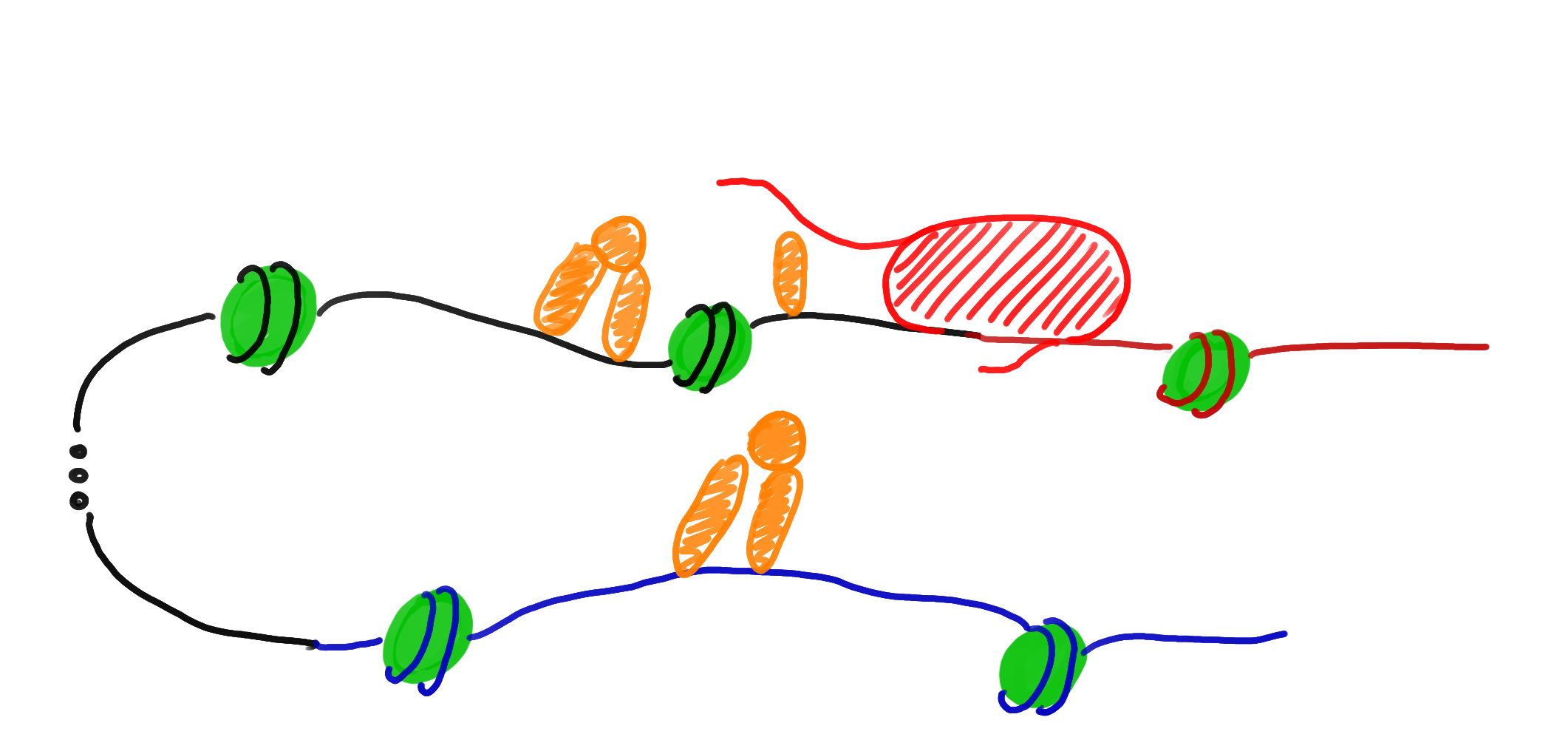
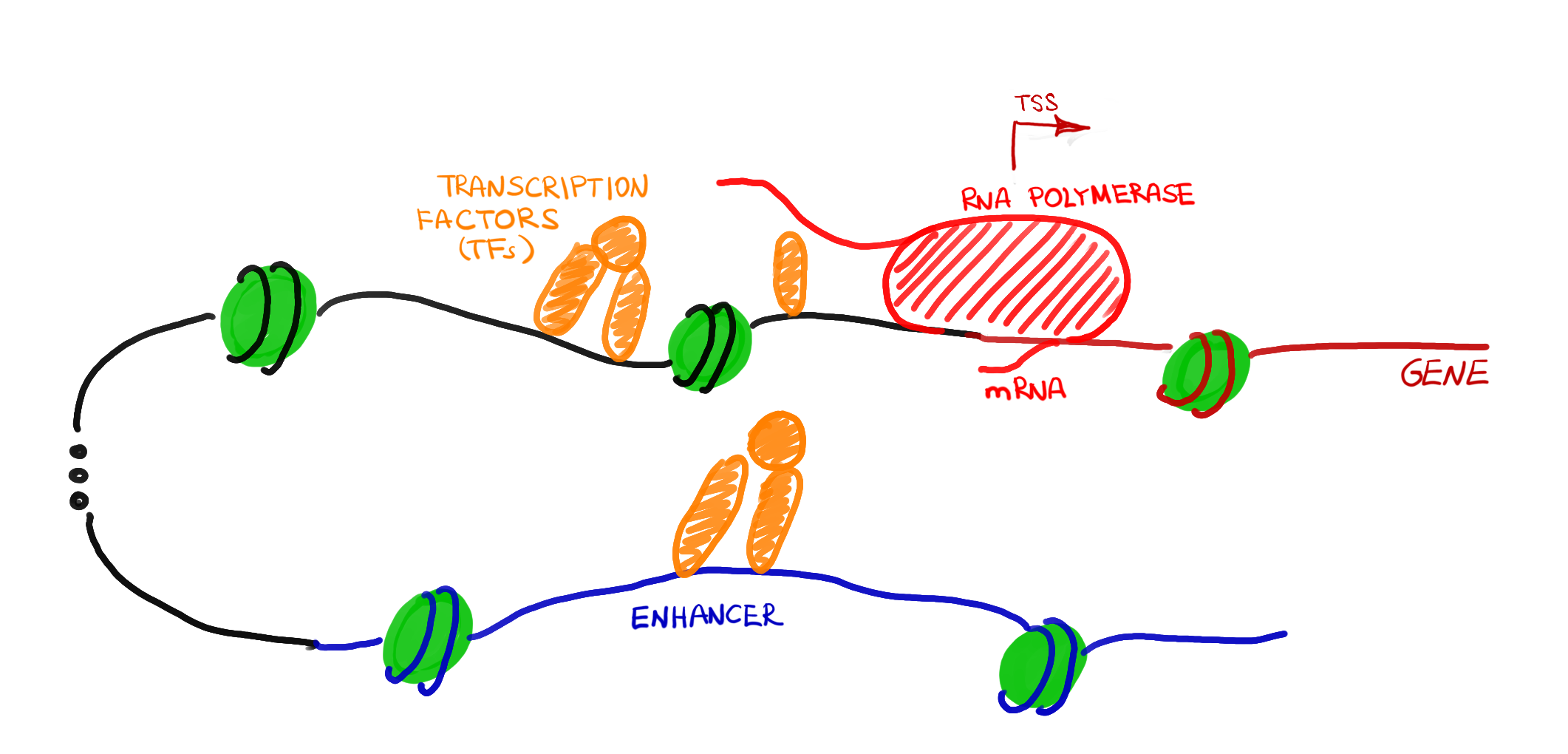
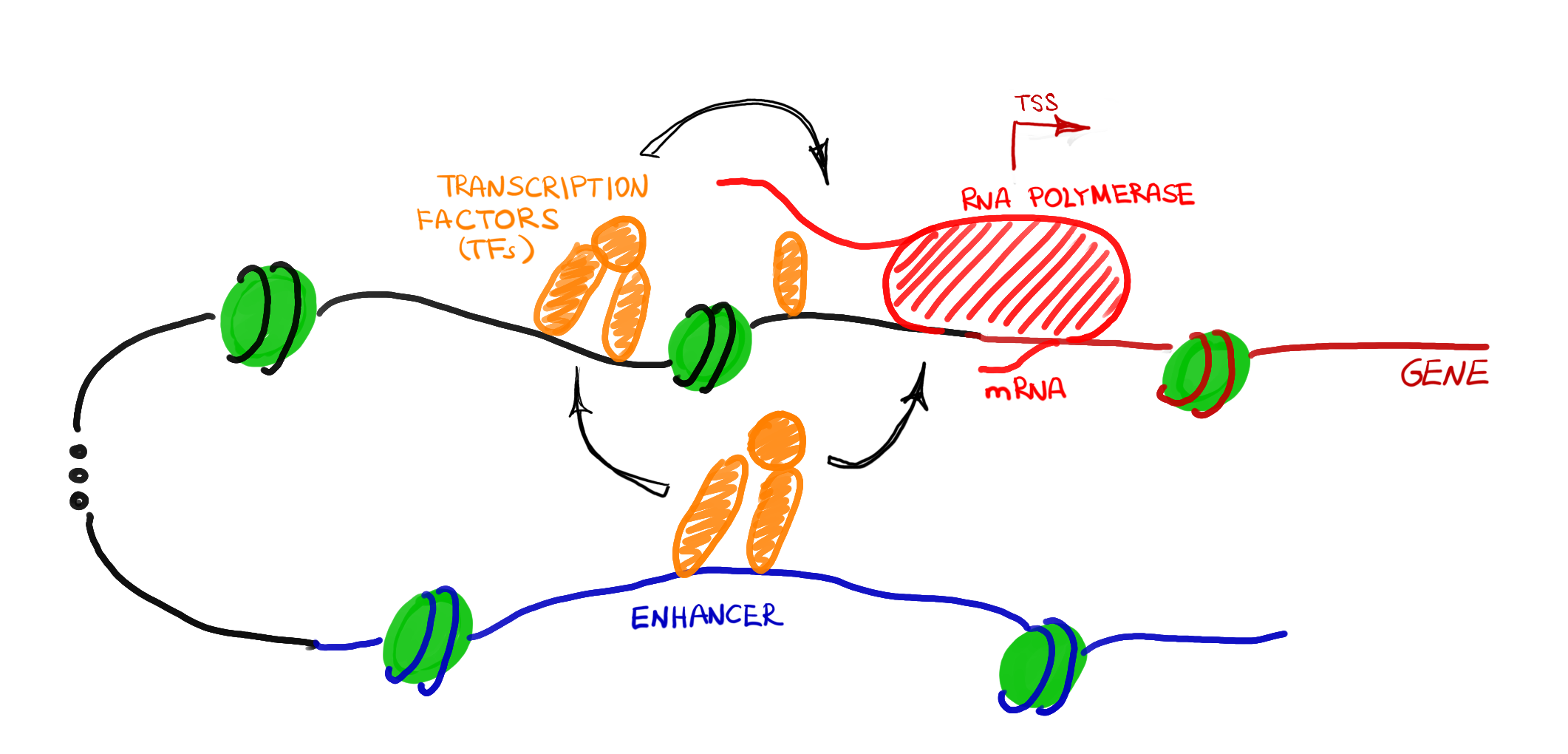
Transcriptional regulation in eukaryotes is
complex.
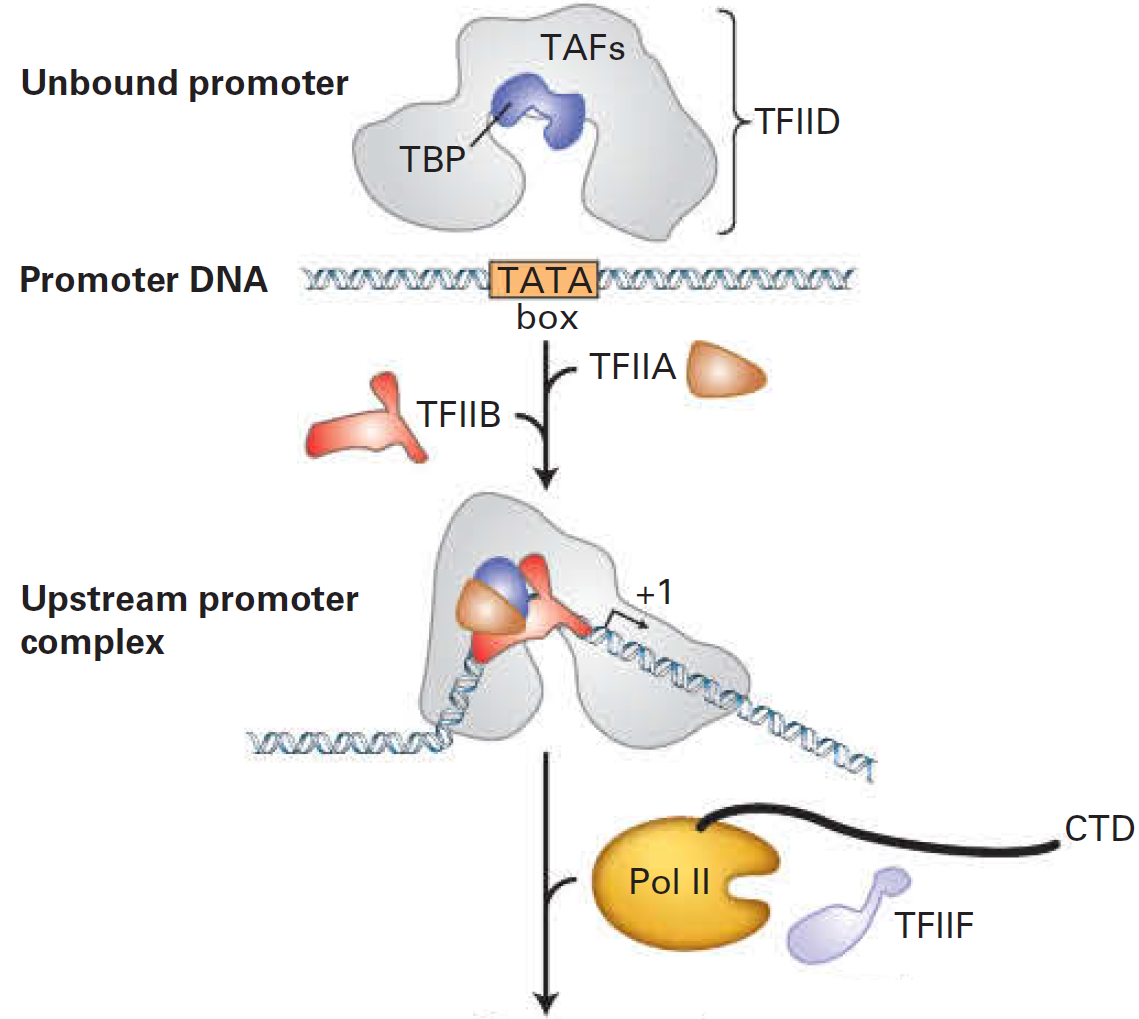 Lodish et al., Molecular Cell Biology, 8th edition, 2016
Lodish et al., Molecular Cell Biology, 8th edition, 2016


"Goldberg-Hogness box"
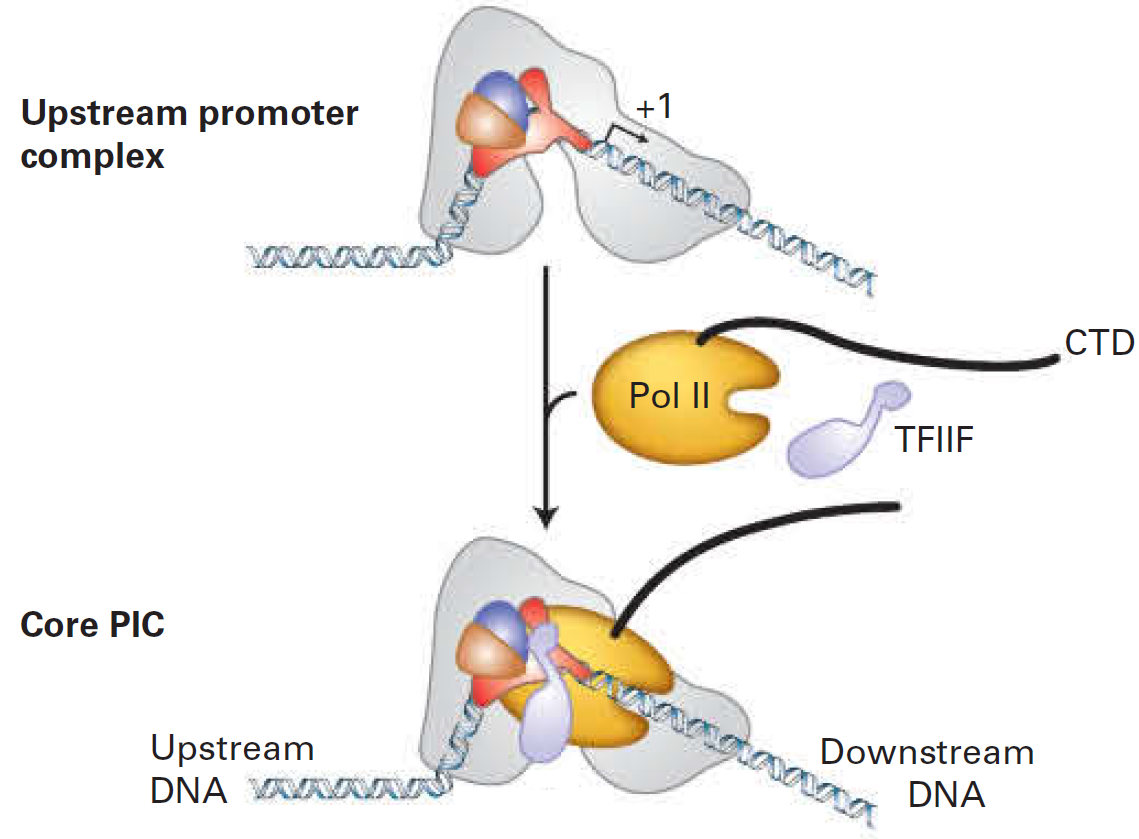
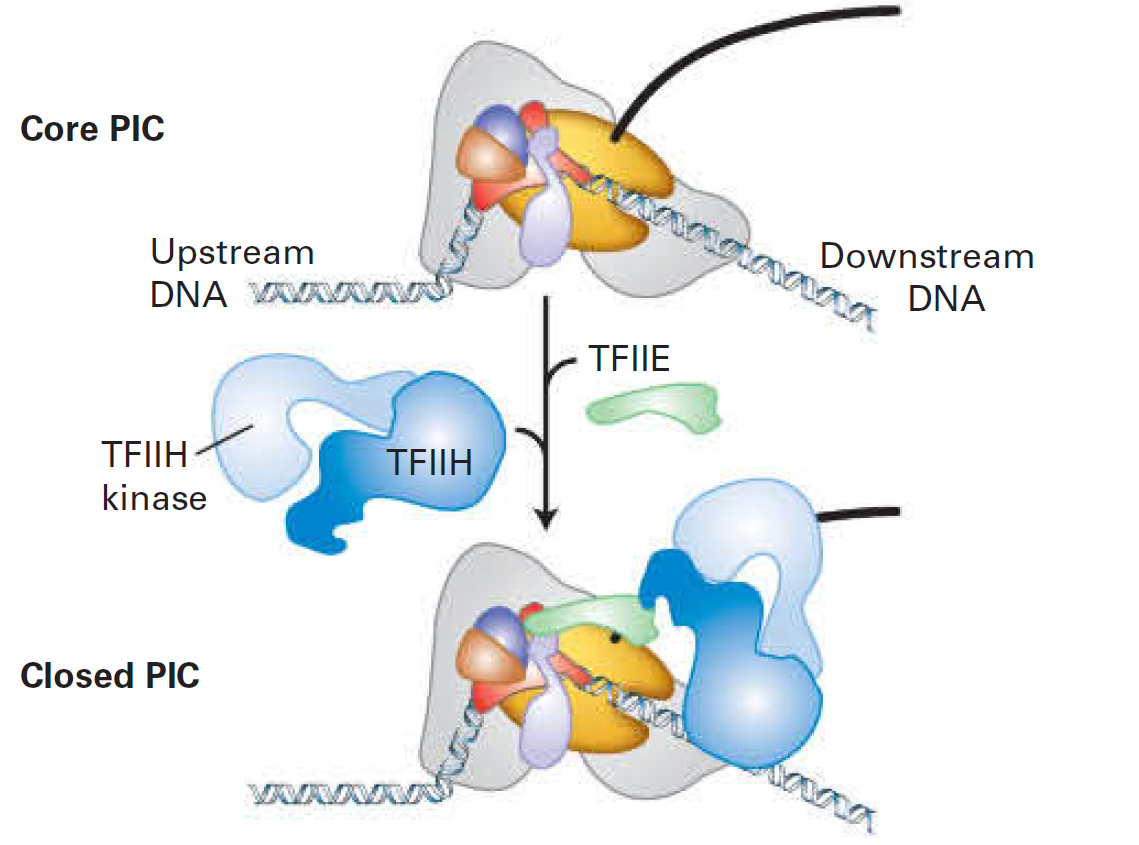
Инициация транскрипции у эукариот


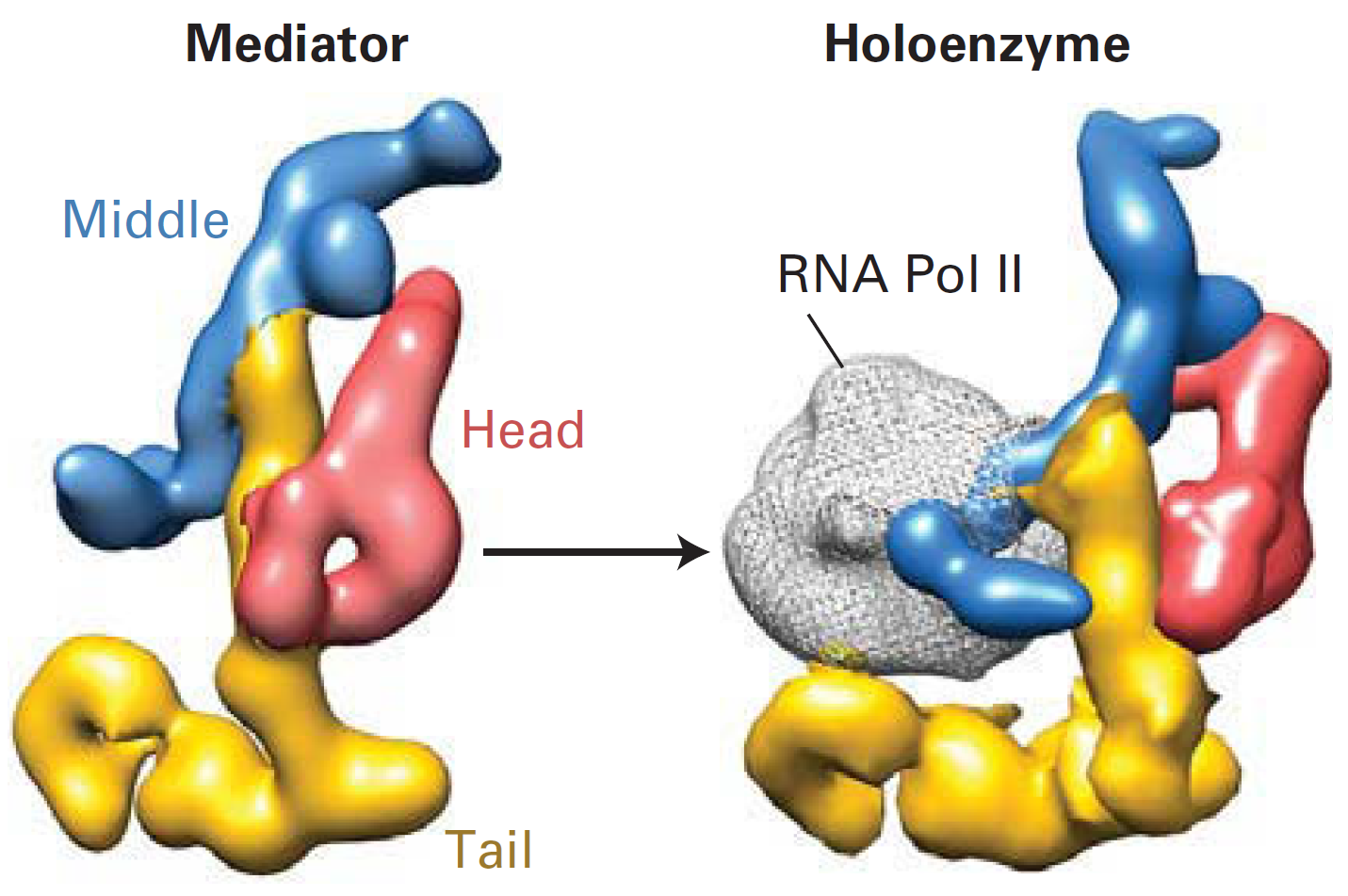
Медиатор и запетливание
Типы промоторов
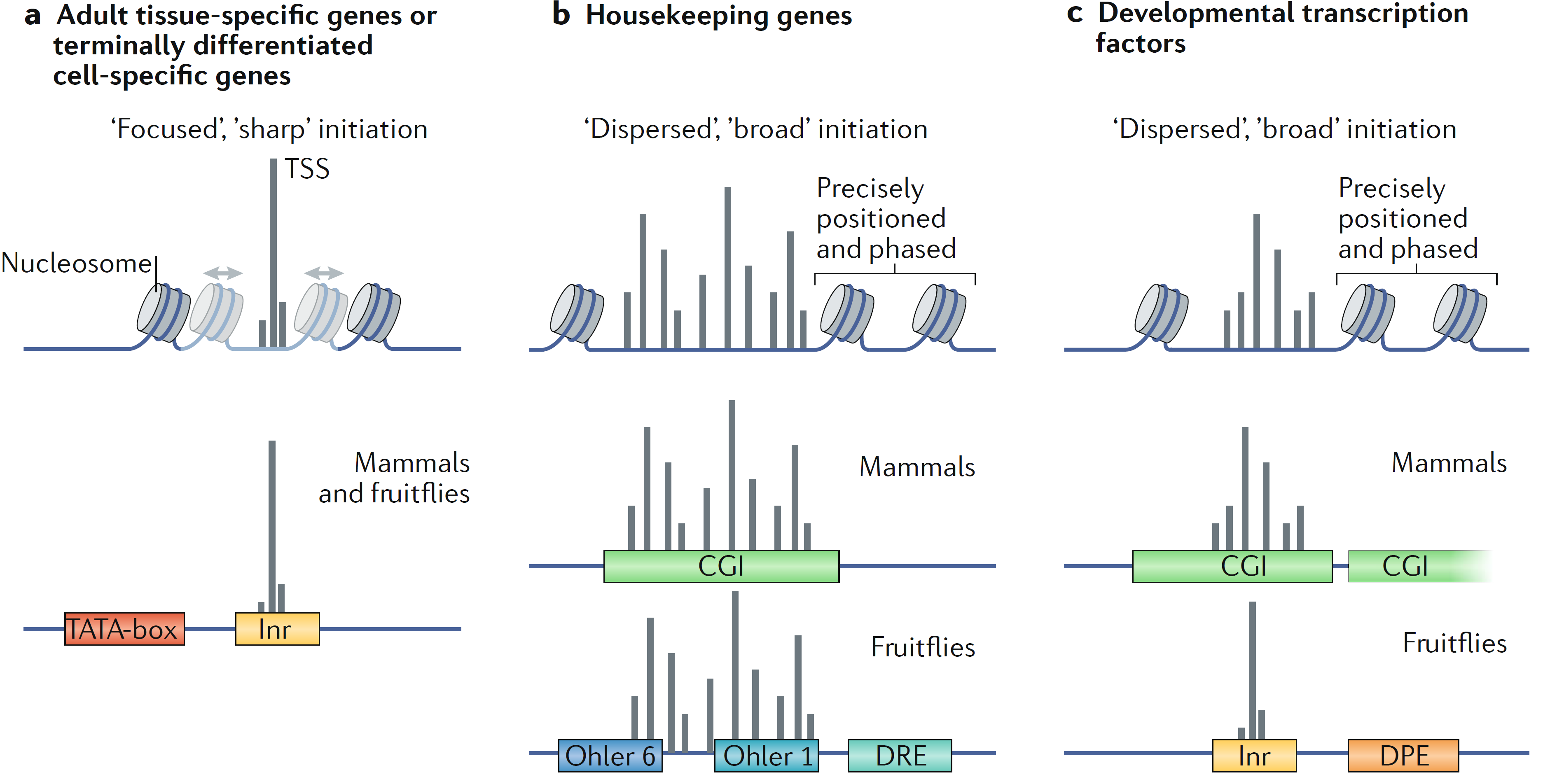
Haberle and Stark, Nature Reviews, 2018
Типы энхансеров
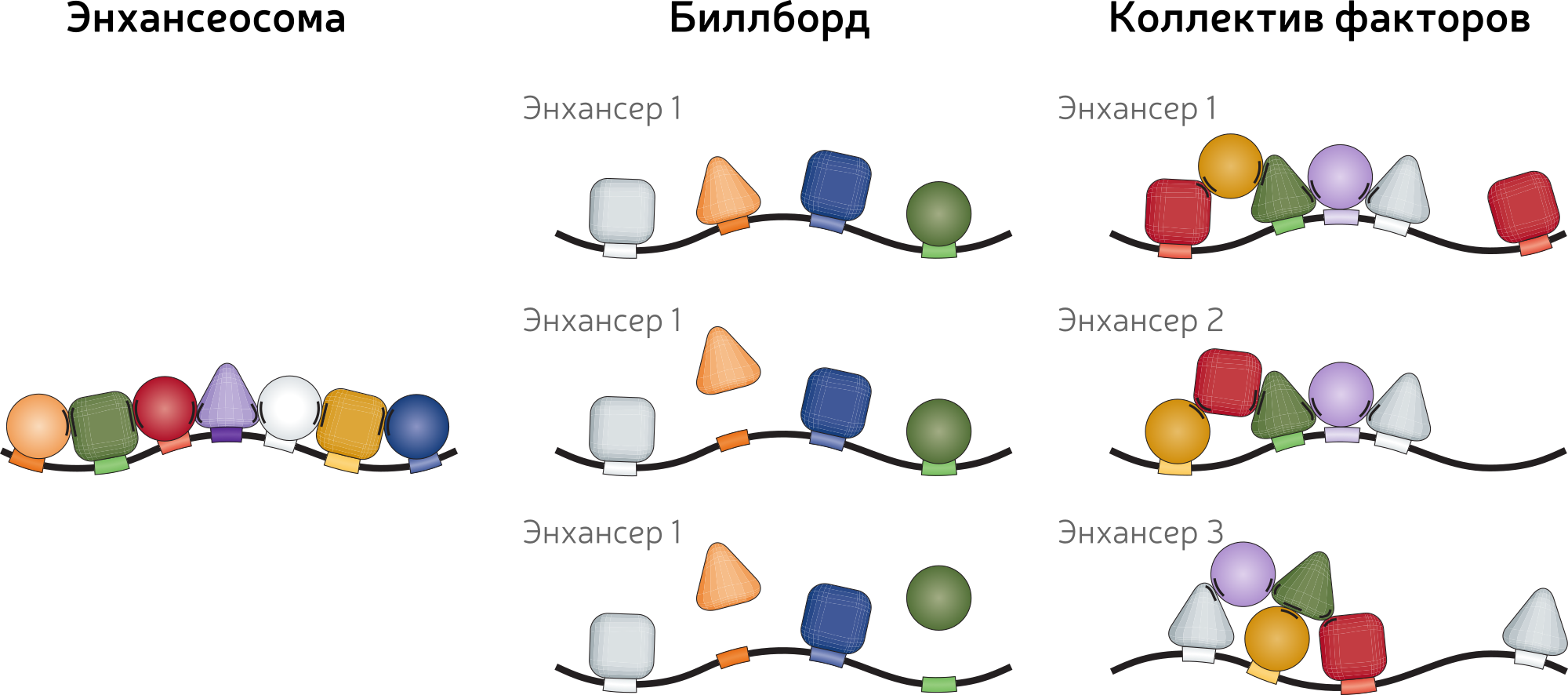
Spitz and Furlong, Nature Reviews Genetics, 2012
Энхансер это тоже промотор
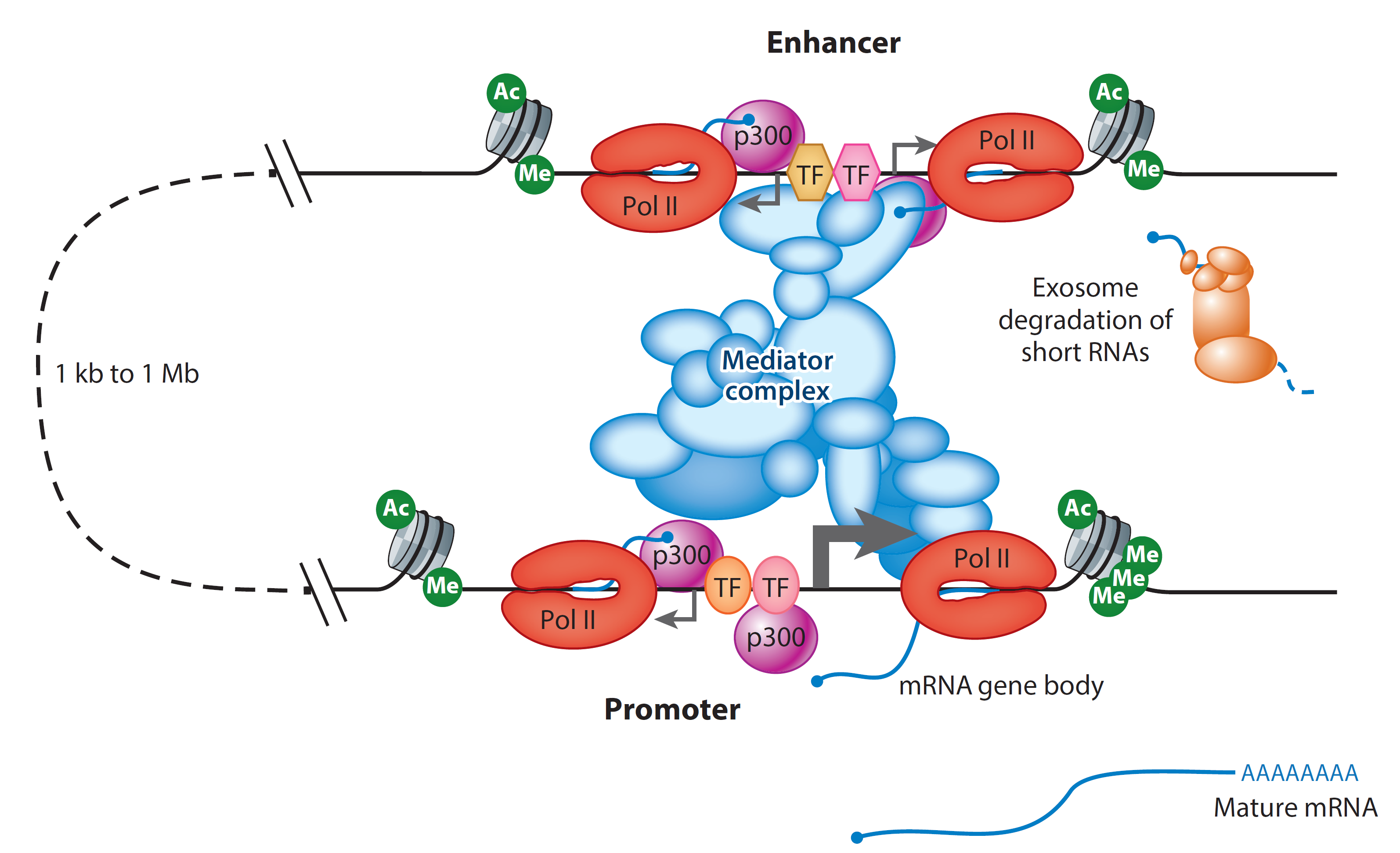
Field and Adelman, Annual Review of Biochemistry, 2020
Зачем транскрибировать энхансер?

Field and Adelman, Annual Review of Biochemistry, 2020
 J. Cooper, The Cell: A Molecular Approach, 8th edition, 2019
J. Cooper, The Cell: A Molecular Approach, 8th edition, 2019
Транскрипционные фабрики
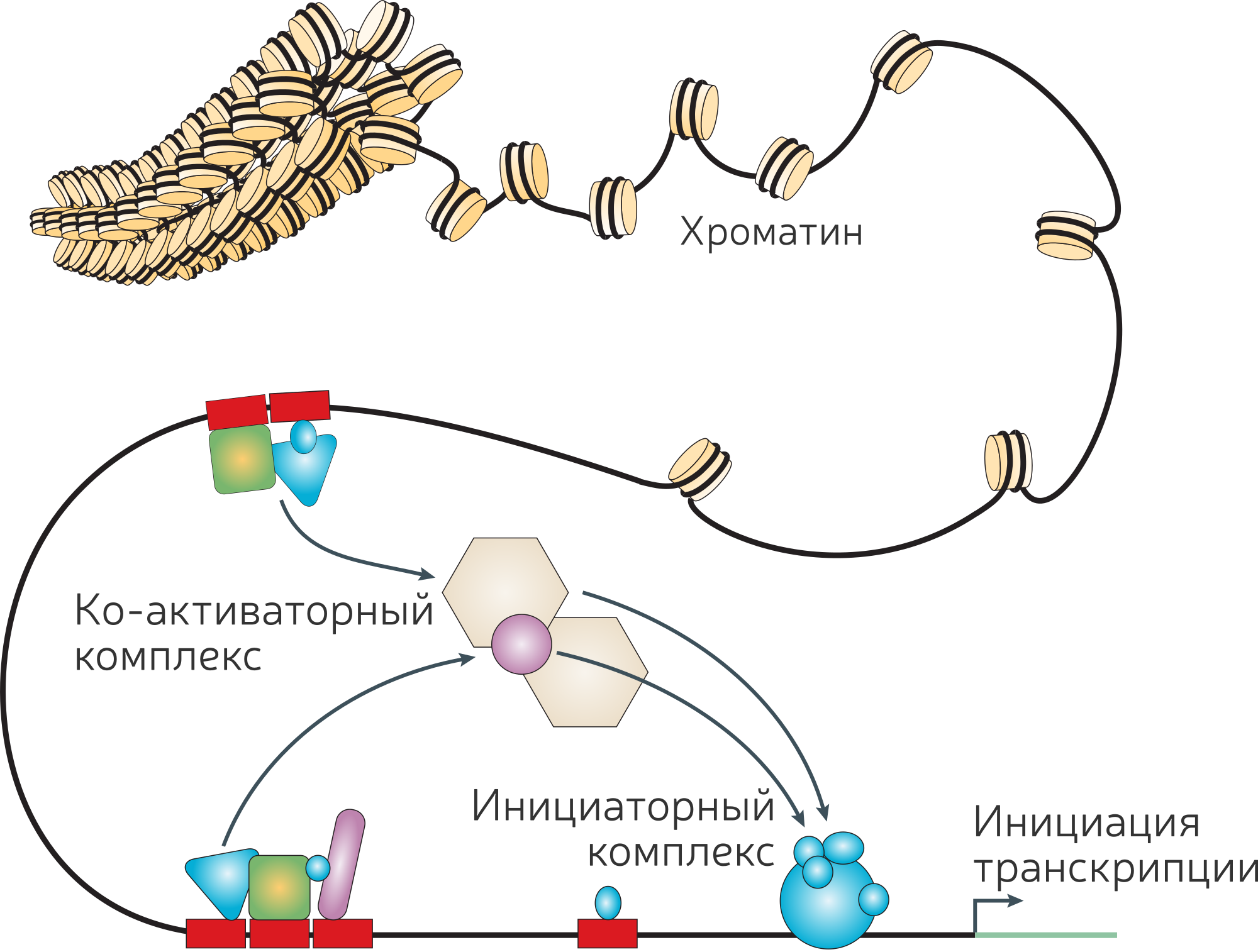
Wassterman, 2004
Где находятся общие и где специализированные факторы транскрипции?
Какие интереснее изучать?


Нобелевская премия по физиологии и медицине 2012
за получение индуцированных плюрипотентных стволовых клеток.
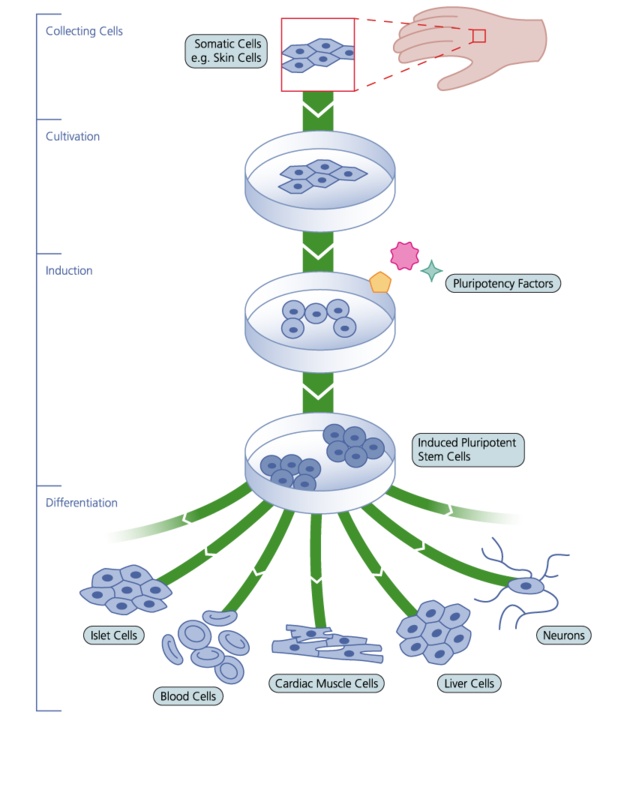 Stammzellnetzwerk.NRW
Stammzellnetzwerk.NRW
Как устроен фактор транскрипции?
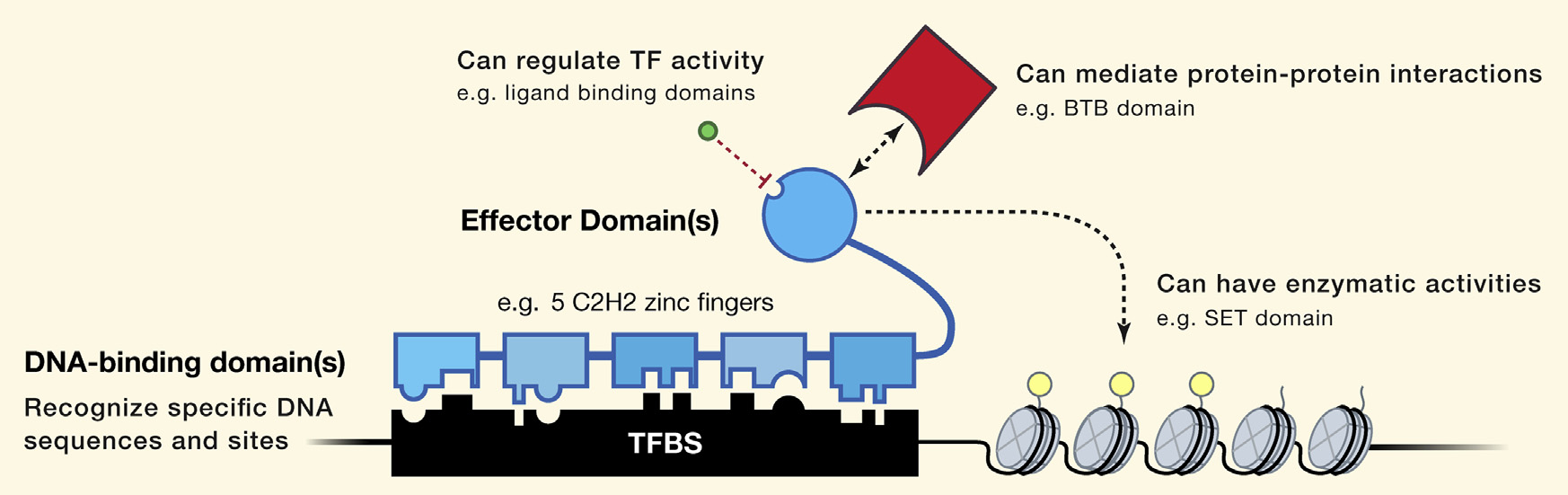 Lambert et al., Cell, 2018
Lambert et al., Cell, 2018
Сколько факторов транскрипции кодируется в геноме человека?
Что такое фактор транскрипции?


dbTF: DNA-binding transcription factor that regulate transcription through the recognition of the genomic address of its target genes.
Результат работы консорциума: курировано 1457 фактора транскрипции (состояние на ноябрь 2020),
из которых 1439 аннотированы в UniProt и HGNC:
http://ebi.ac.uk/QuickGO/targetset/dbTF
ДНК-связывающий домен определяет специфичность
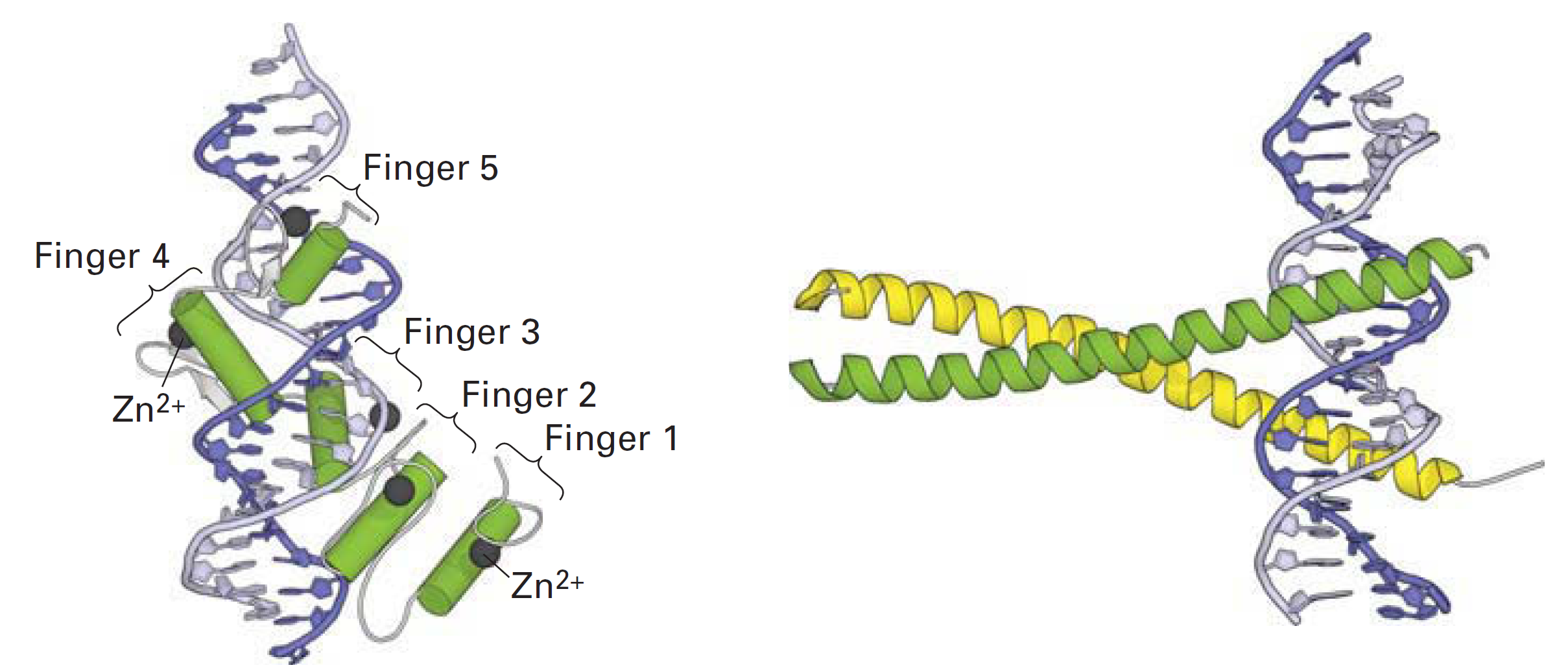 Lodish et al., Molecular Cell Biology, 8th edition, 2016
Lodish et al., Molecular Cell Biology, 8th edition, 2016
TFClass: наиболее подробная классификация факторов транскрипции человека и других млекопитающих по структурам ДНК-связывающих доменов
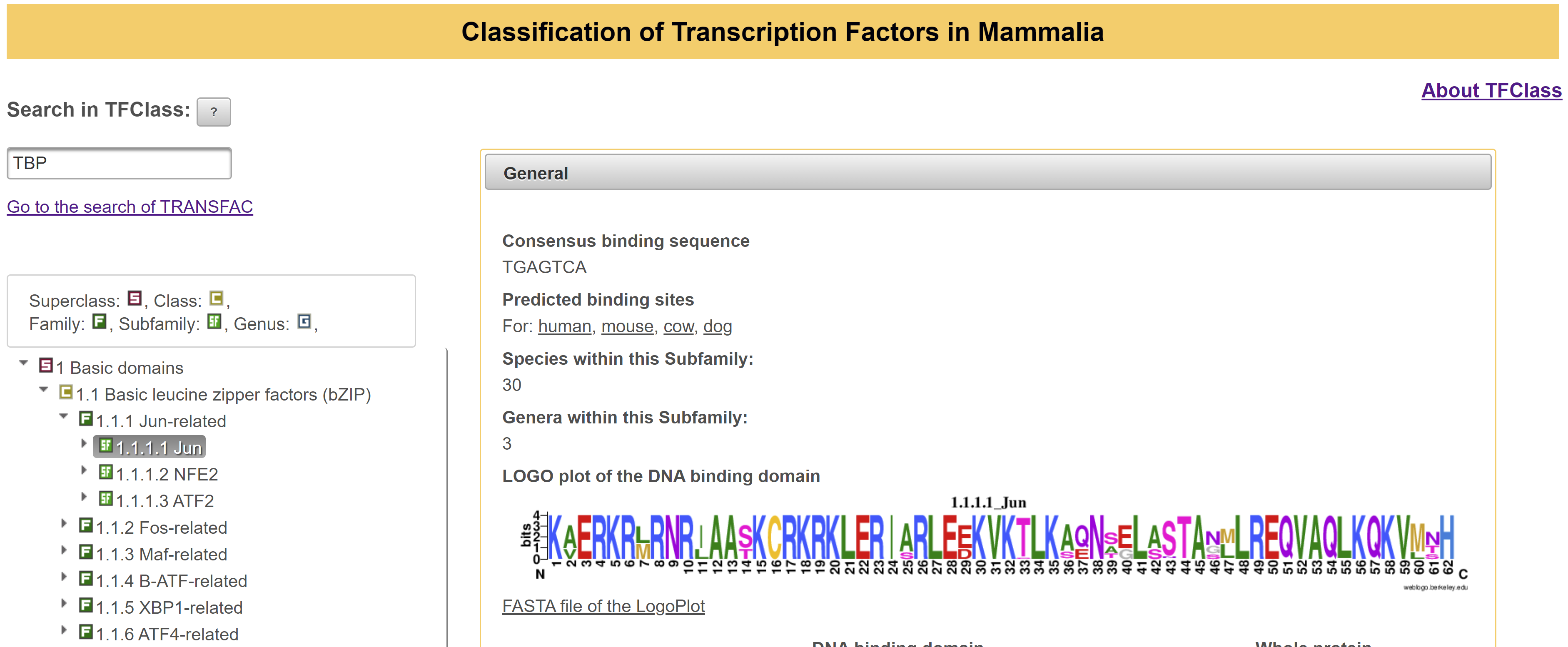 Wingender et al., Nucleic Acids Research, 2018
Wingender et al., Nucleic Acids Research, 2018
Энхансеосома энхансера интерферона-бета
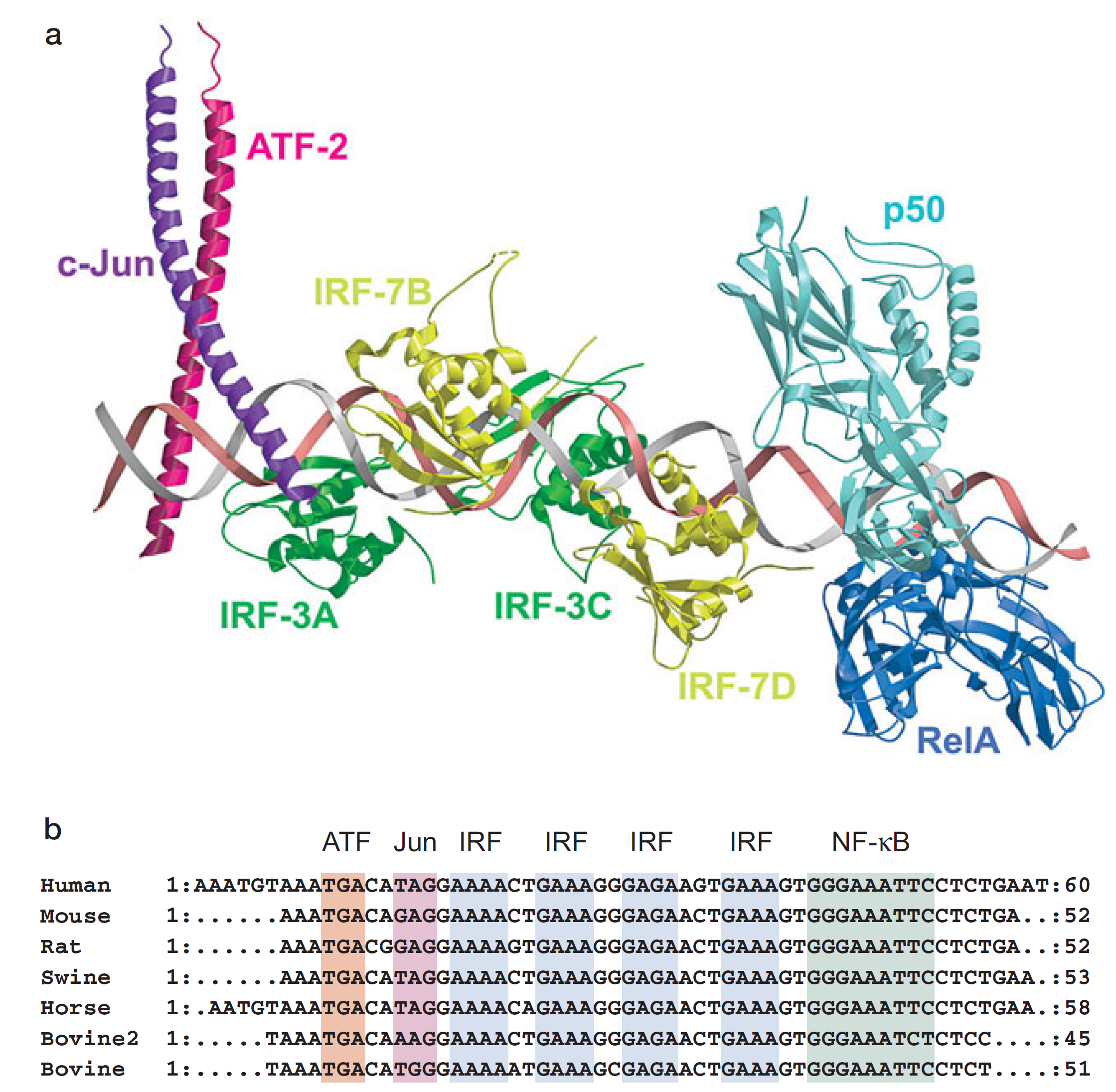 Watson et al., Molecular Biology of the Gene, 7th edition, 2014
Watson et al., Molecular Biology of the Gene, 7th edition, 2014
Комбинаторная регуляция транскрипции и петлевая модель
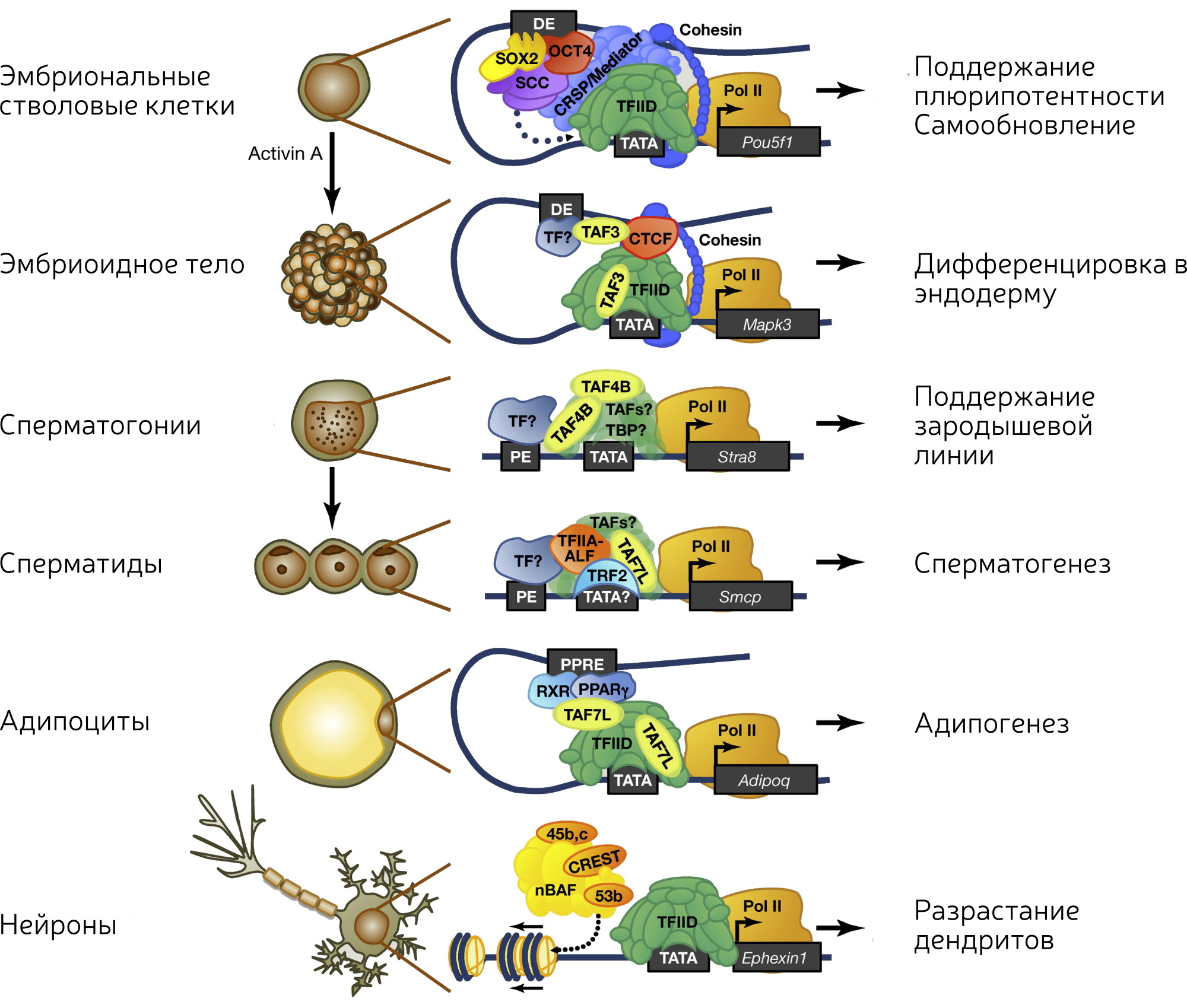 Looping back to leap forward: Transcription enters a new era, Cell, 2014
Looping back to leap forward: Transcription enters a new era, Cell, 2014
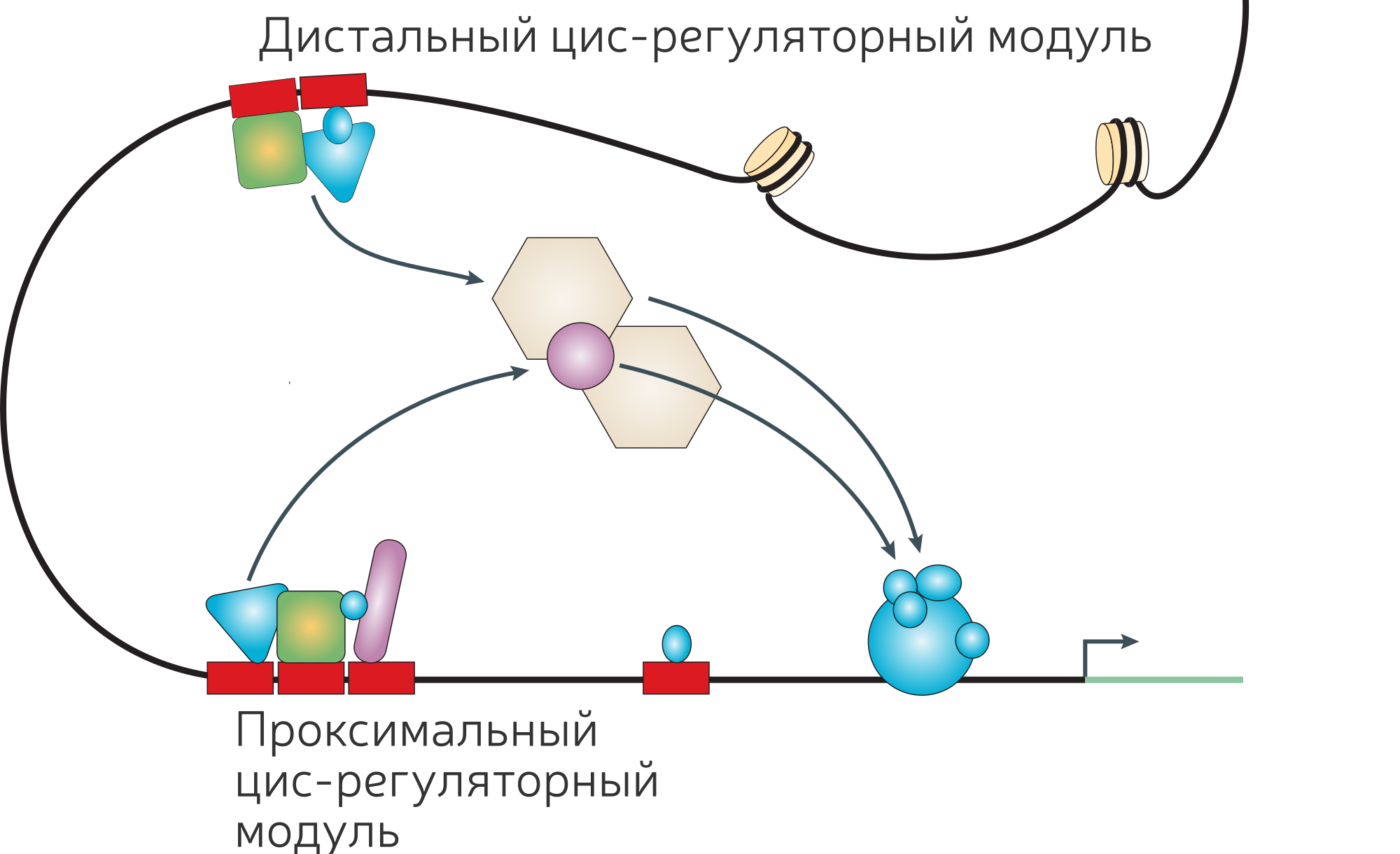
Wasserman, 2004
Почему цис-регуляторные регионы (районы),
элементы и модули?
Цис-транс-тест (тест на комплементацию)
 Benzer S, 1957, The elementary units of heredity; Lewis E B, 1951, Pseudoallelism and gene evolution
Benzer S, 1957, The elementary units of heredity; Lewis E B, 1951, Pseudoallelism and gene evolution
Трансвекция у плодовых мушек
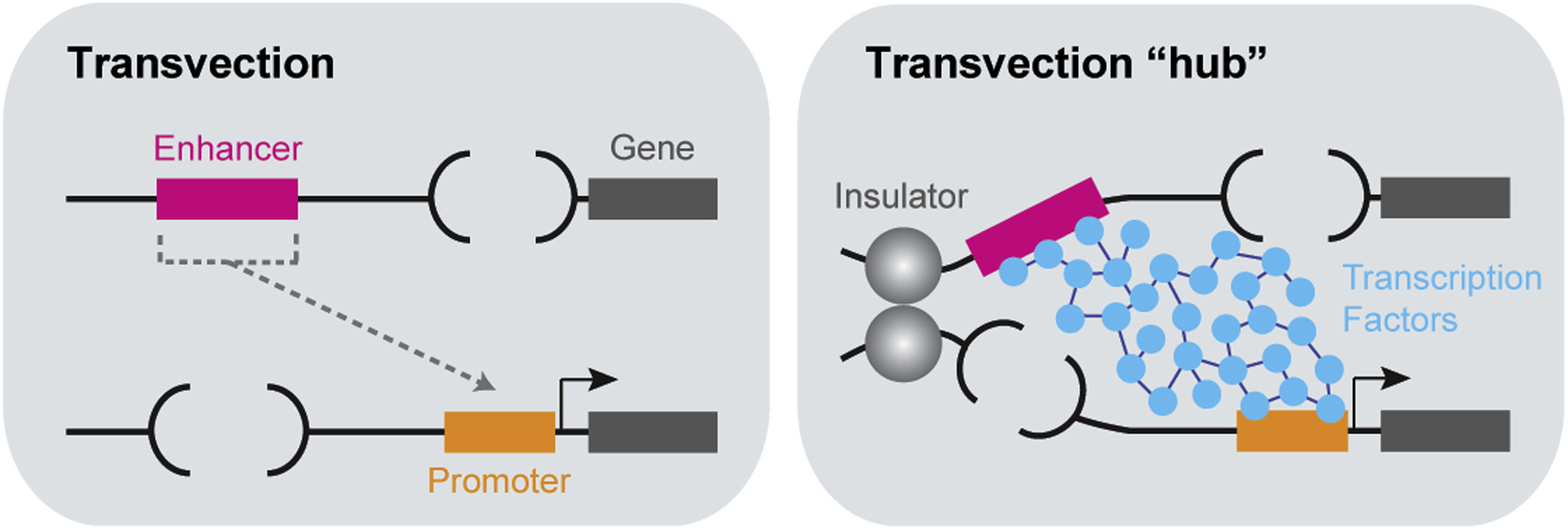 Рисунок из Tsai et al., Molecular Cell, 2018; Эффект обнаружен E.B. Lewis, Am. Nat., 1954
Рисунок из Tsai et al., Molecular Cell, 2018; Эффект обнаружен E.B. Lewis, Am. Nat., 1954
Проект FANTOM5

Кэп-анализ экспрессии генов - версия HeliScopeCAGE
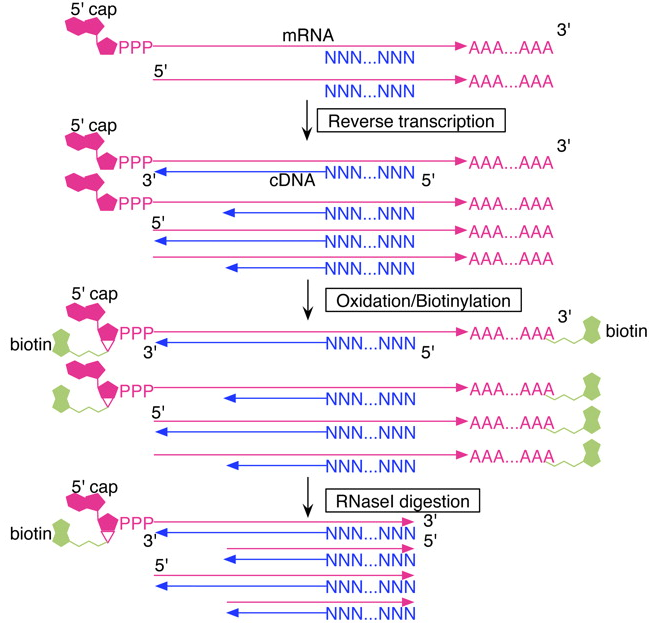
Kanamori-Katayama et al., Genome Res, 2011
Кэп-анализ экспрессии генов
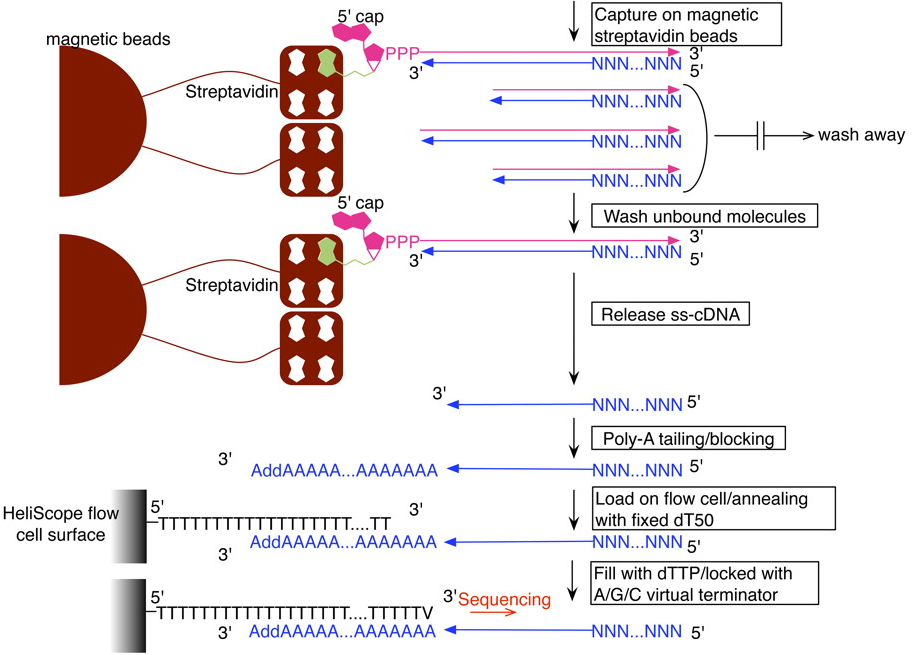
Kanamori-Katayama et al., Genome Res, 2011
Helicos - высокопроизводительное секвенирование без ПЦР
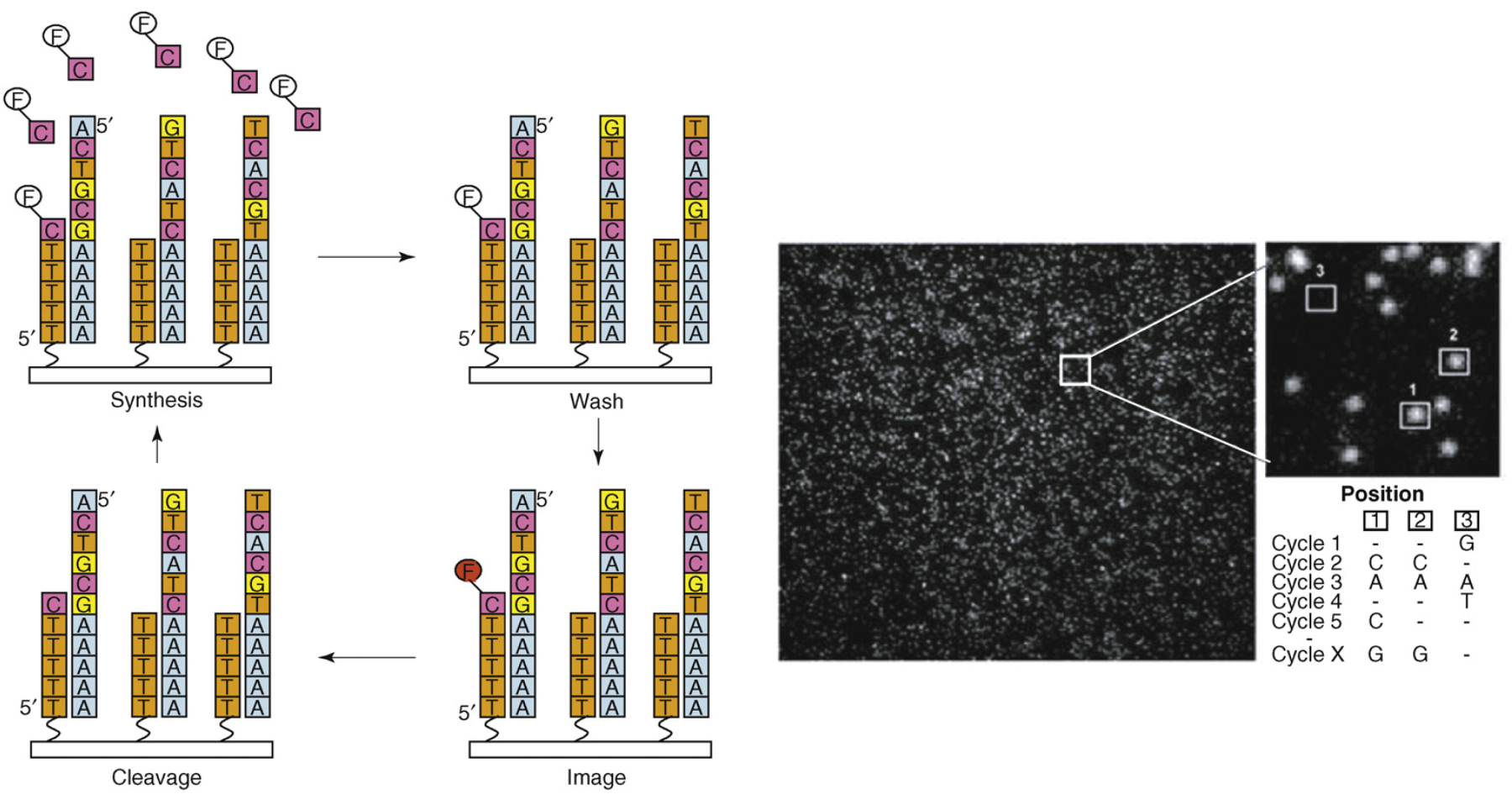
P.K. Gupta, Trends in Biotechnology, 2008
Анализ промотерома человека и мыши
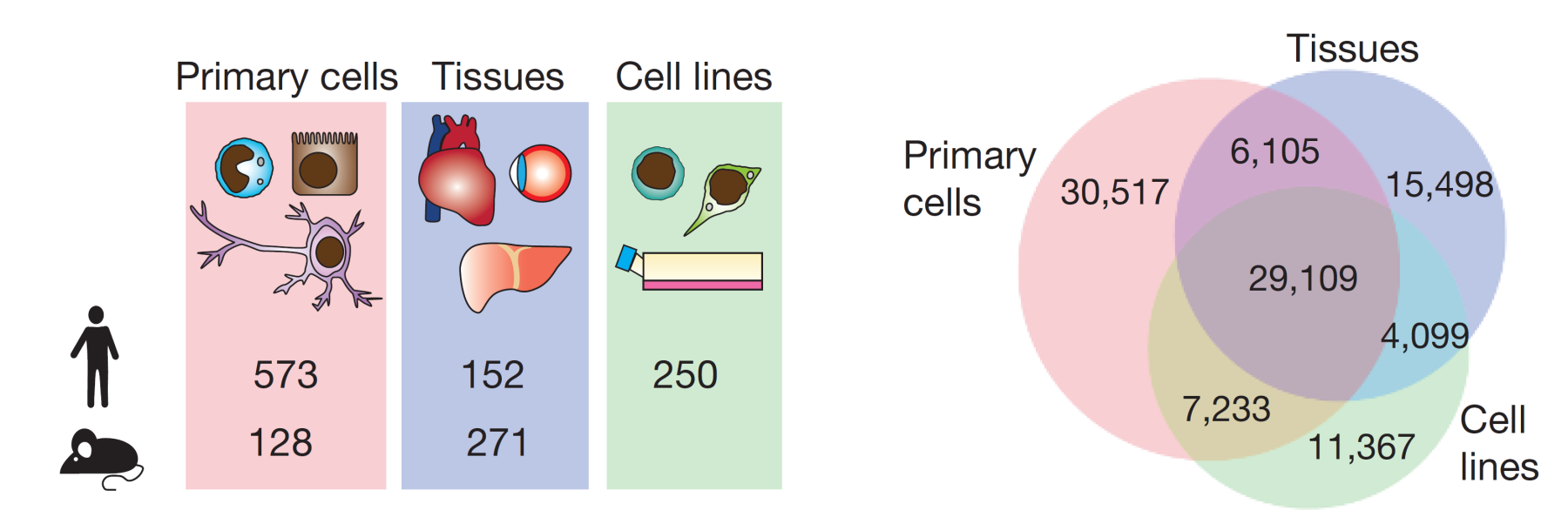
Forrest et al., Nature, 2014
Дифференциальная активность промотеров
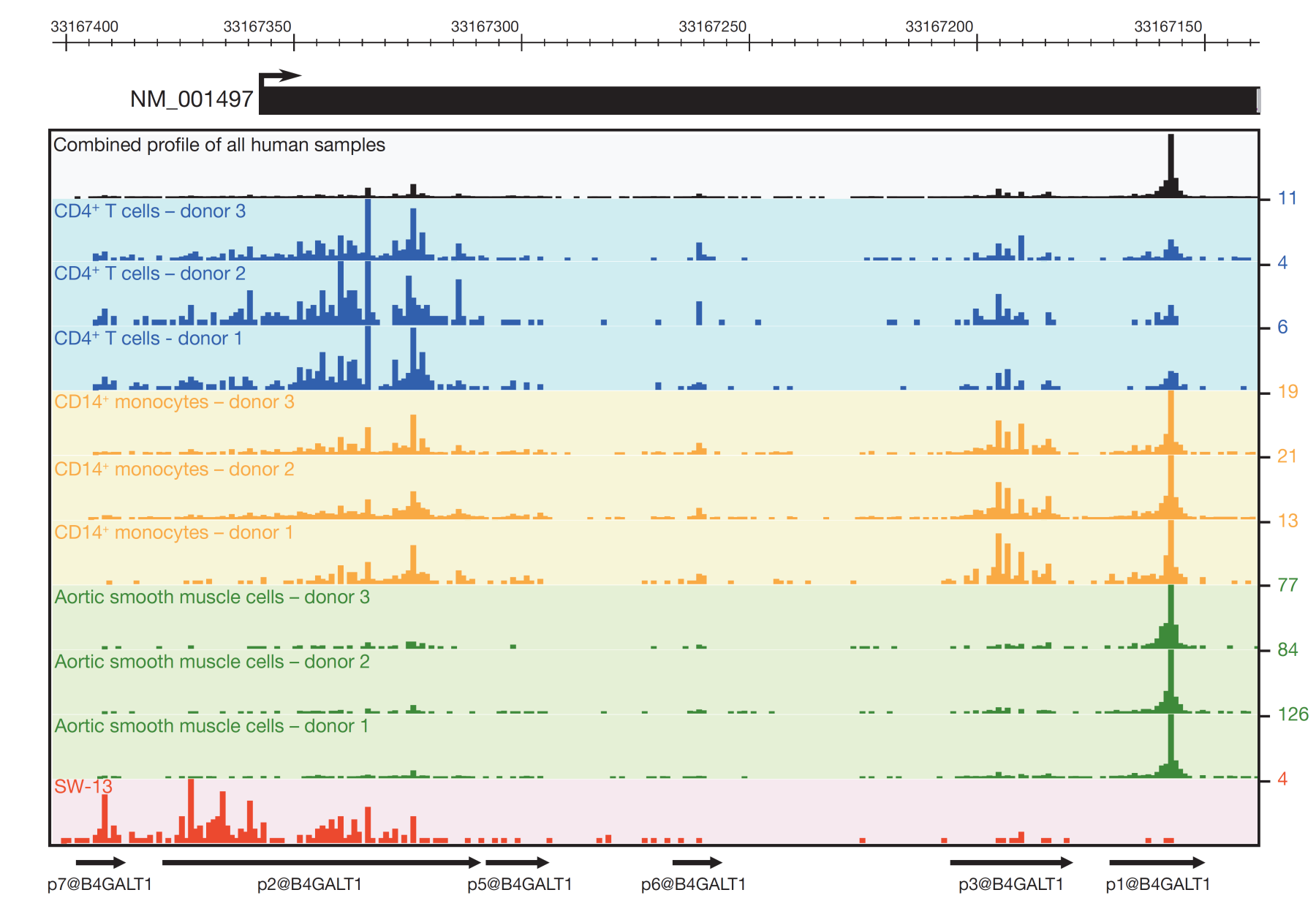
Forrest et al., Nature, 2014
Достоверная идентификация ткань-специфичных промотеров и промотеров домашнего хозяйства
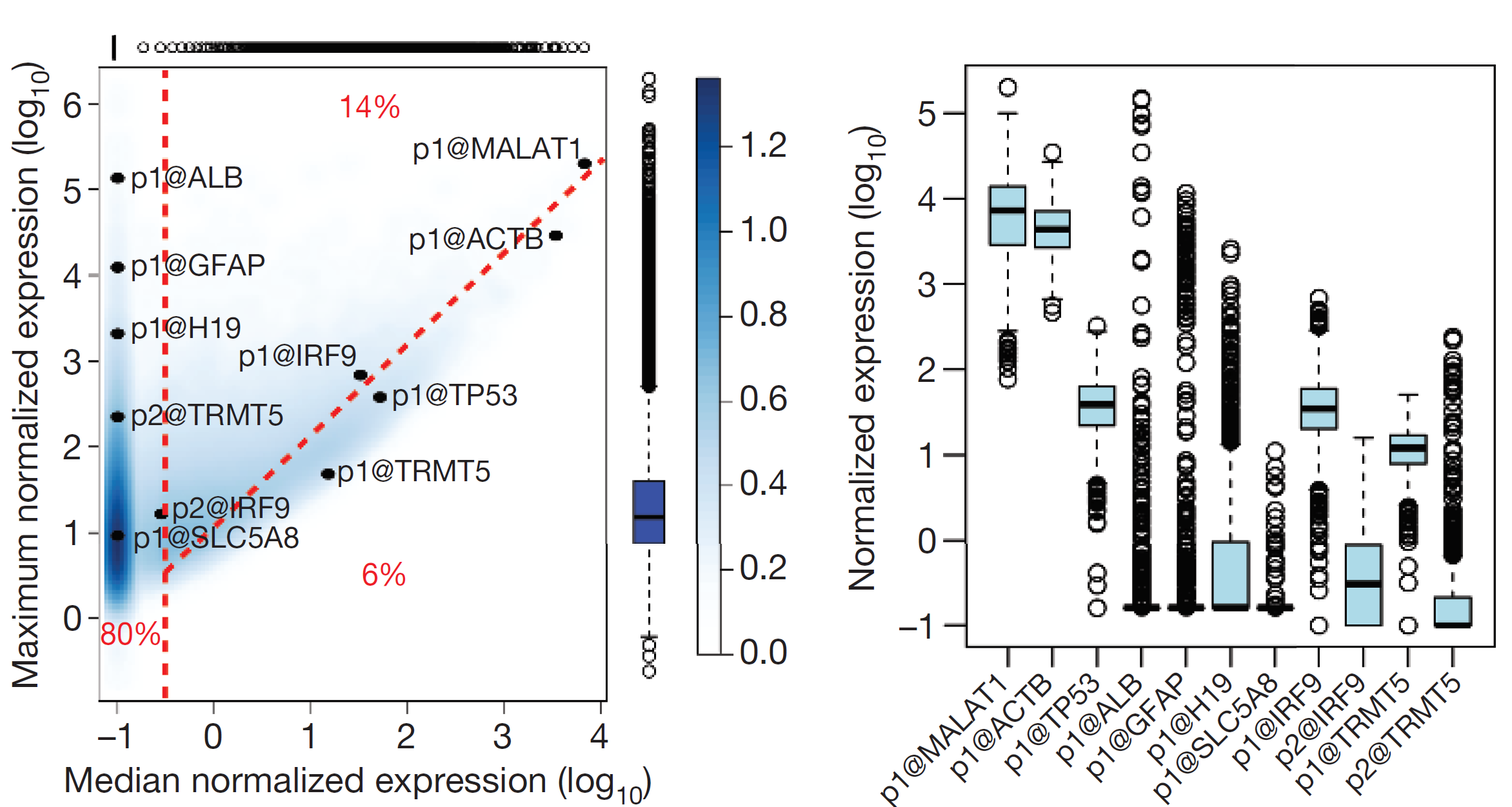
Forrest et al., Nature, 2014
Упрощенная схема коэкспрессии промотеров

Трансдифференцировка
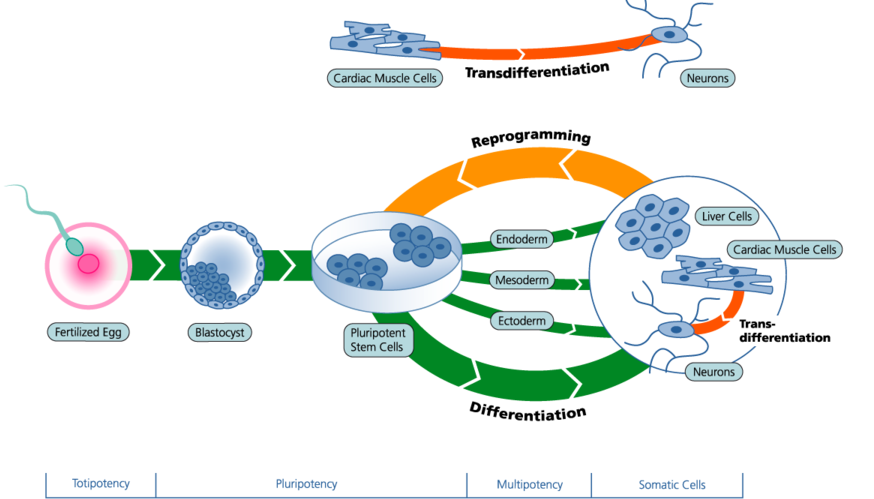 Stammzellnetzwerk.NRW
Stammzellnetzwerk.NRW
Перепрограммирование клеток с помощью факторов транскрипции и эпифакторов
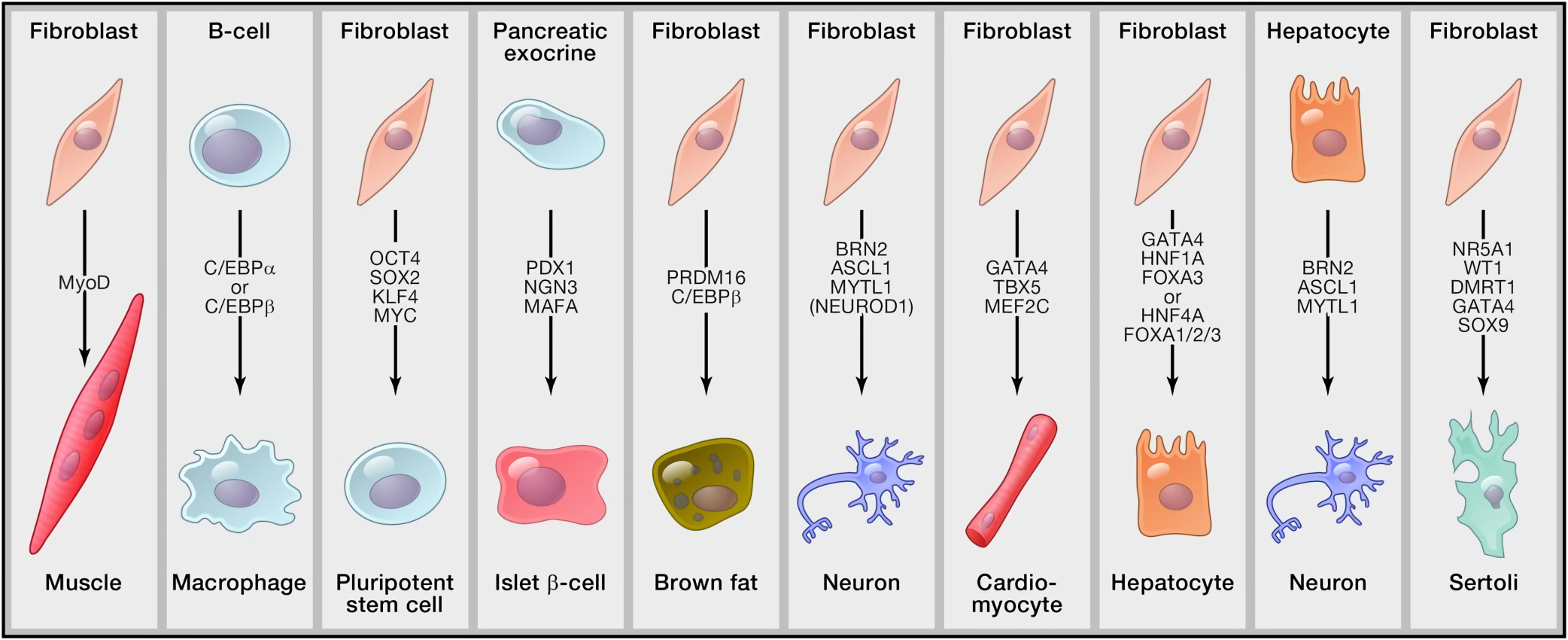 Transcriptional Regulation and Its Misregulation in Disease,
Transcriptional Regulation and Its Misregulation in Disease, Lee and Young, Cell, 2013
Mogrify: direct cellular reprogramming
 Rackham et al., Nature Genetics, 2016
Rackham et al., Nature Genetics, 2016
 Rackham et al., Nature Genetics, 2016
Rackham et al., Nature Genetics, 2016
Почему еще интересно изучать уровень транскрипции?
Геномные варианты в регуляторных последовательностях и их связь с фенотипами
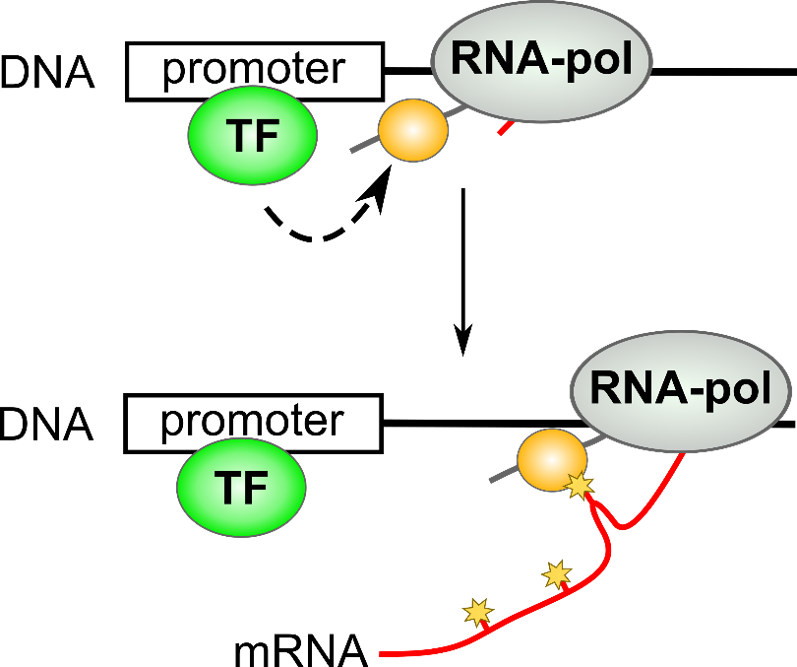
Пост-транскрипционная регуляция экспрессии
Transcription therefore is not only a catalyst of mRNA synthesis but also provides a platform that enables imprinting, which coordinates between transcription and mRNA decay.
Haimovich et al., Biochim Biophys Acta, 2013
Promoter sequences direct cytoplasmic localization and translation of mRNAs during starvation in yeast.
Zid and O'Shea, Nature, 2014
Ко-транскрипционное связывание РНК-связывающих белков
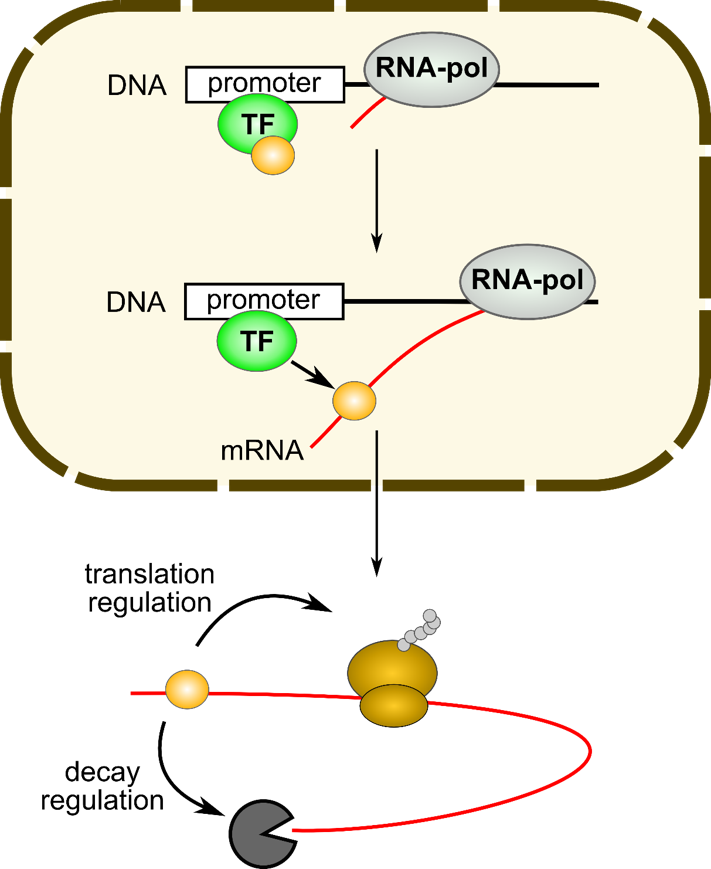
Ко-транскрипционная модификация РНК - "эпитранскриптомика"
Супермутанты среди нас
 Alessio Lonigro
Alessio Lonigro
Супермутанты среди нас

Найдено 13 больных-но-здоровых человек, носителей мутаций, но не соответствующих им моногенных заболеваний: Муковисцидоз, Синдром Смита-Лемли-Опица, Семейная дизавтономия, Простой буллезный эпидермолиз, Синдром Пфайффера, Аутоиммунный полиэндокринный синдром 1 типа, Акампомелическая кампомелическая дисплазия, Ателостеогенез
Take-home

Транскрипция определяет клеточную идентичность
Специфичность транскрипции определяется специализированными факторами транскрипции которые находят свой "геномный адрес" с помощью ДНК-связывающих доменов
Старт (сайт инициации) транскрипции это не точка, а район
Транскрипция это не только транскрипция