Транскрипция,
ее регуляция,
и как ее изучают
(с помощью секвенирования и биоинформатики)
Ваня Кулаковский, Пущино-Москва
Часть 3: мотивы ДНК-белкового узнавания
и как их опознать

Регуляторная сеть генной экспрессии
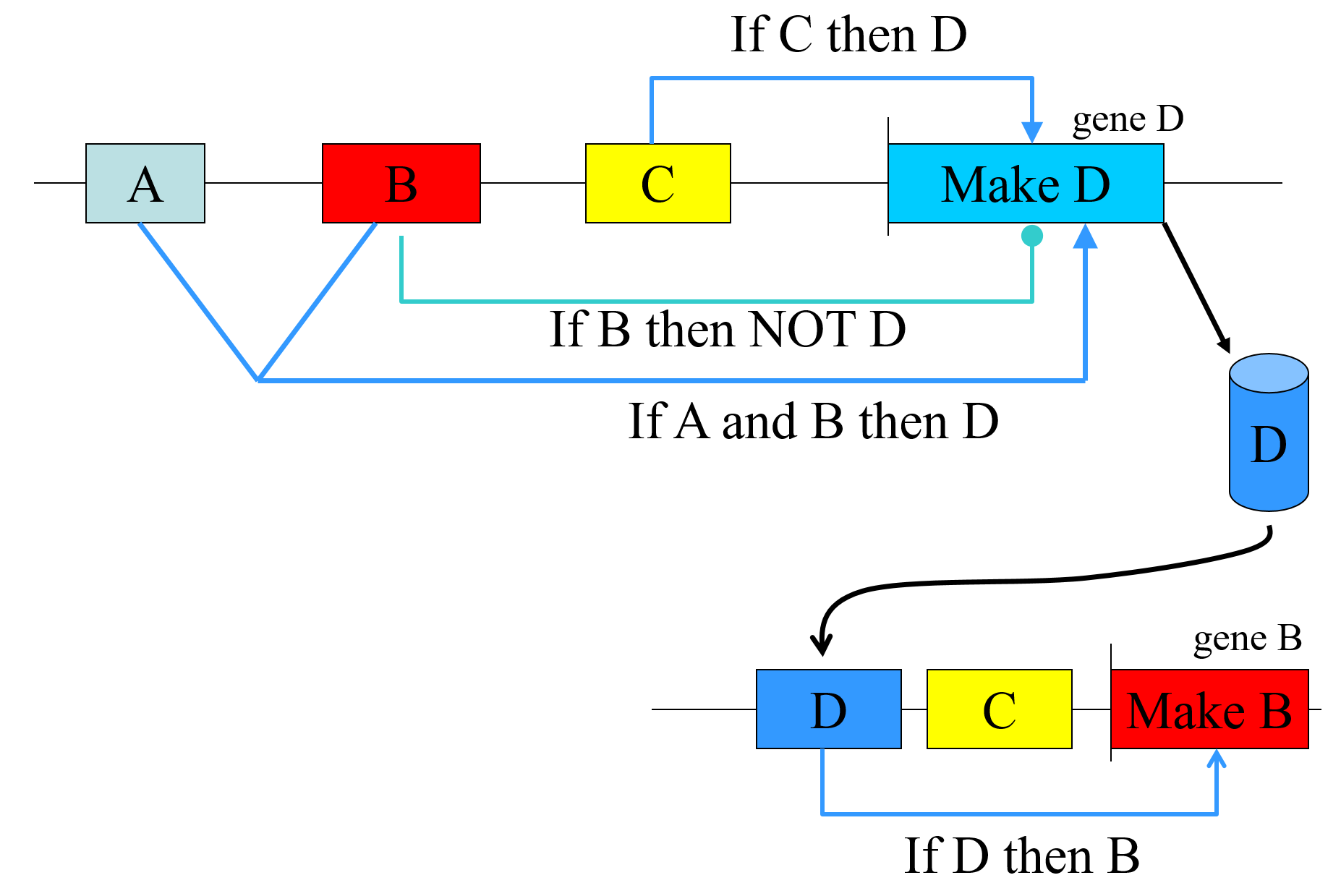 Jianhua Ruan, http://www.cs.utsa.edu/~jruan/teaching/cs5263_spring_2017/index.htm
Jianhua Ruan, http://www.cs.utsa.edu/~jruan/teaching/cs5263_spring_2017/index.htm
От связывания факторов транскрипции
к системной биологии


Как фактор транскрипции находит свой геномный адрес?
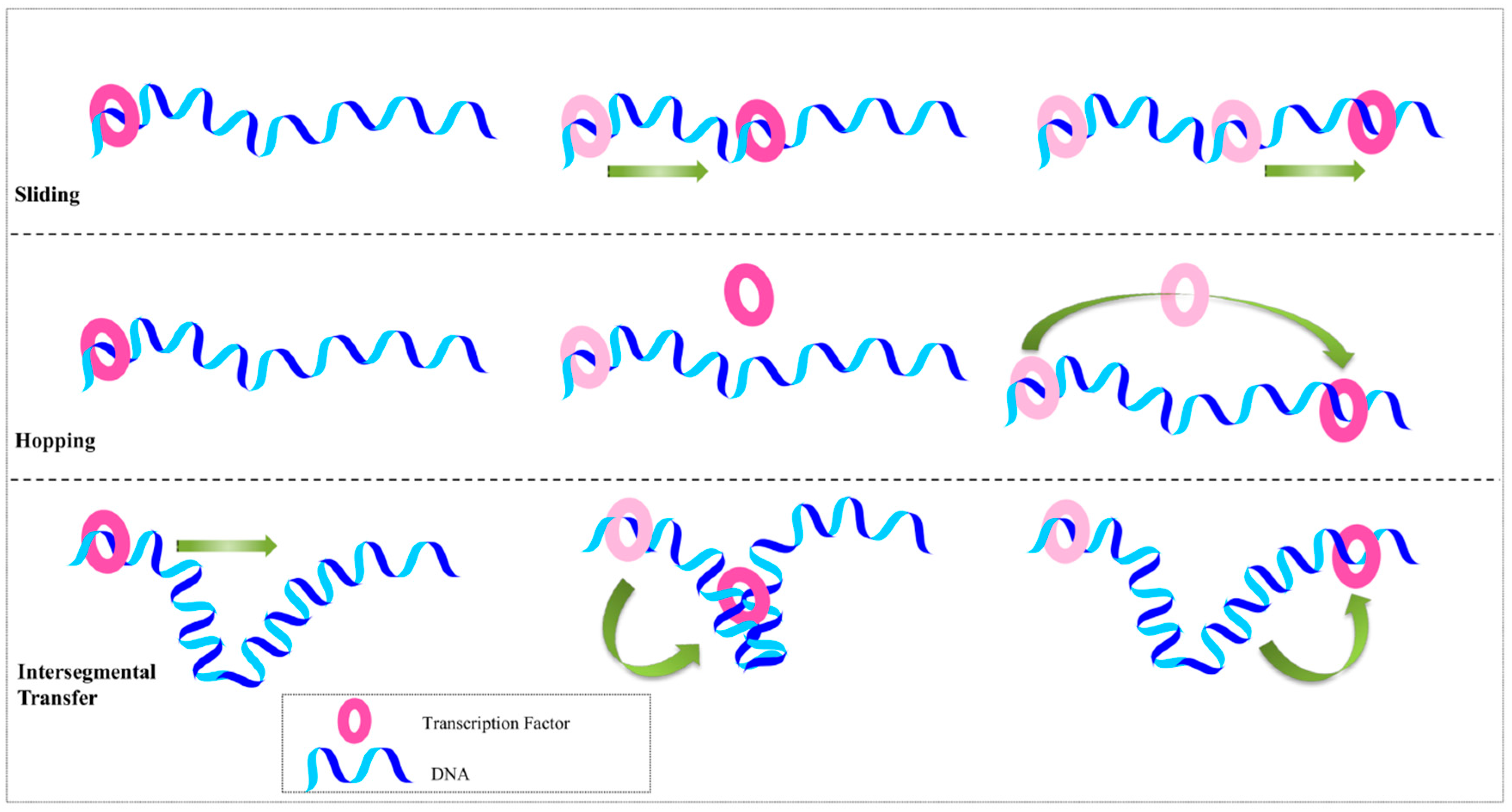 Yesudhas et al., Genes, 2017
Yesudhas et al., Genes, 2017
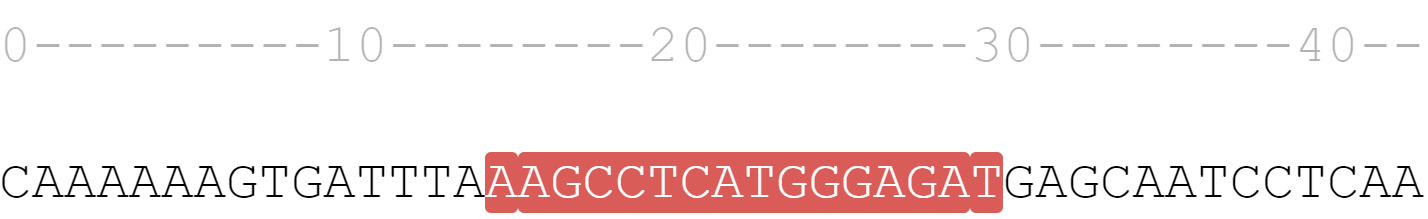
Как нам выяснить геномный адрес связывания?
Как нам уточнить "номер дома"?
Связывание факторов транскрипции определяется последовательностью!
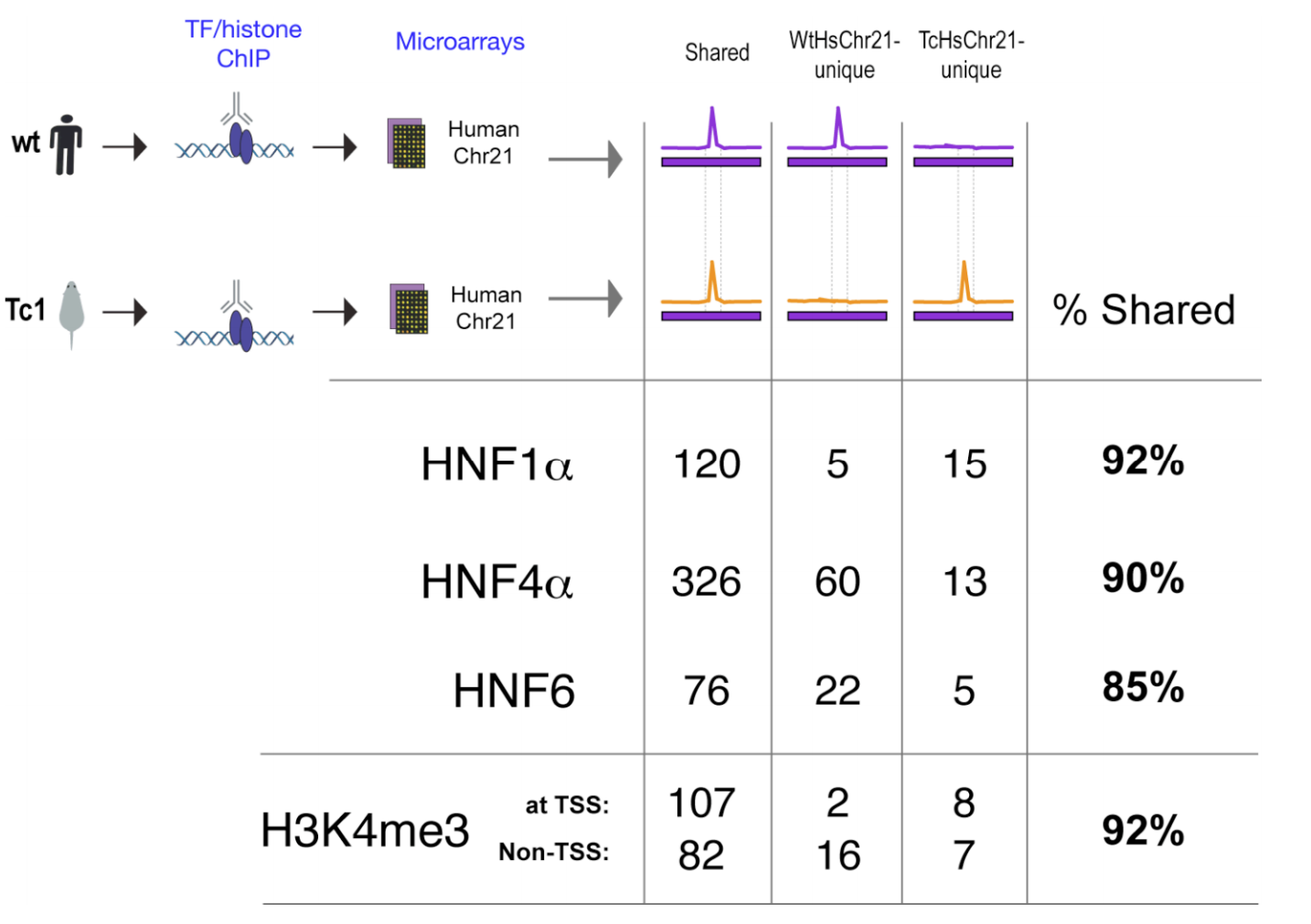
Wilson et al. 2018, Science
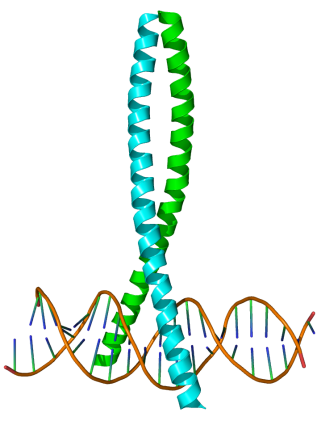
Специфичность определяется структурой

Занегина О.Н., Сравнительная характеристика структур ДНК-белковых комплексов,
диссертация на соискание степени кандидата наук
ДНК-белковое узнавание
 James Q. "Spider" Rich and Homer "Boots" Randolph III, 1963
James Q. "Spider" Rich and Homer "Boots" Randolph III, 1963
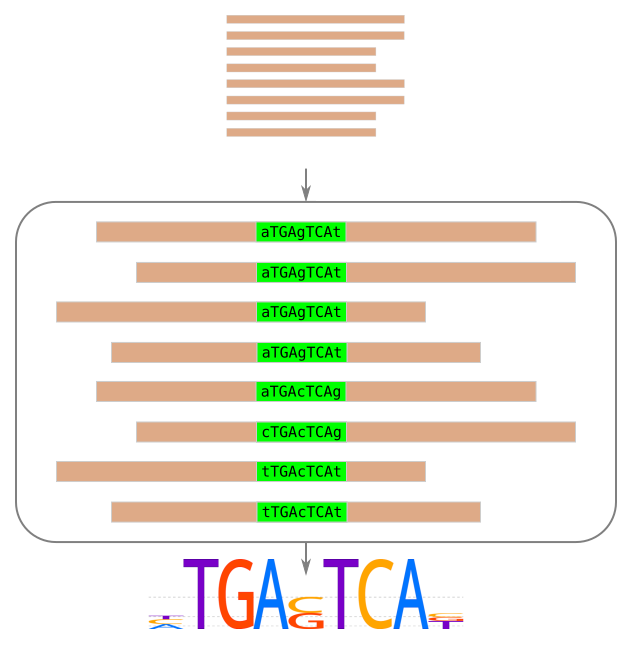
Идентификация мотива - de novo / ab initio motif discovery
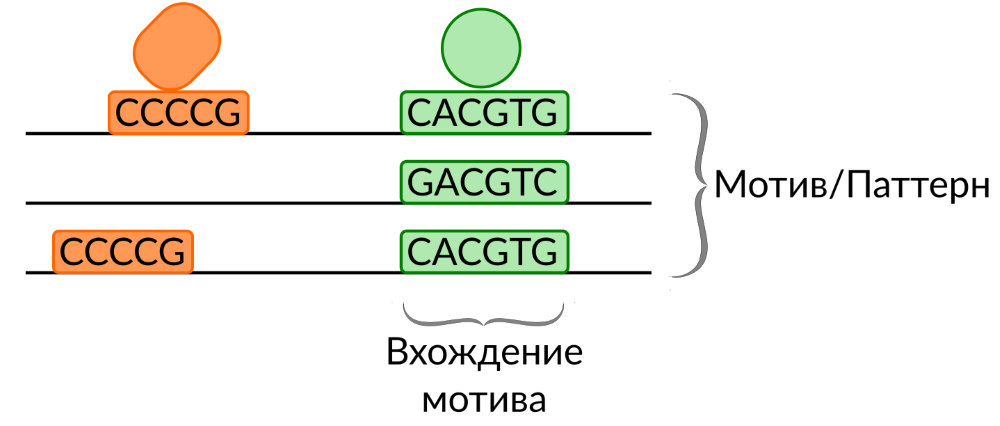
Поиск вхождений - motif finding & pattern matching
"Классическая" постановка задачи идентификации мотива
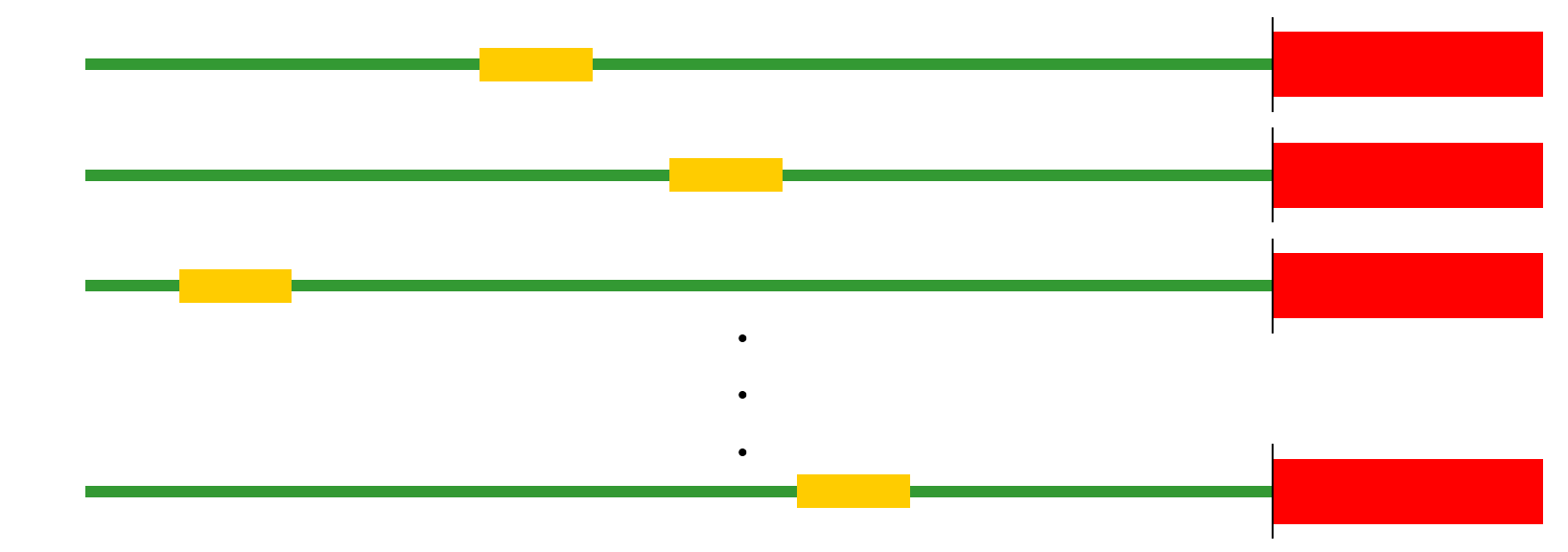
Один промотер - много видов (консервативность регуляции)
Один вид - много промотеров (корегуляция)
Jianhua Ruan, http://www.cs.utsa.edu/~jruan/teaching/cs5263_spring_2017/index.htm
 Watson, Molecular Biology of the Gene, 2014
Watson, Molecular Biology of the Gene, 2014
Прямые методы анализа:
Факт специфического связывания - EMSA
Предпочитаемое место - ДНКазный футпринтинг
Предпочитаемое слово - SELEX
Итого: 10-100 связываемых последовательностей небольшой длины
George Edward Pelham Box
(October 18, 1919 – March 28, 2013)
Essentially, all models are wrong,
but some are useful.
in Empirical Model-Building and Response Surfaces (1987)
Дискретные и вероятностные модели мотива
Список слов, консенсус, регулярное выражение⇩
Позиционно-весовая матрица
⇩
Сложная модель
Позиционно-весовая матрица

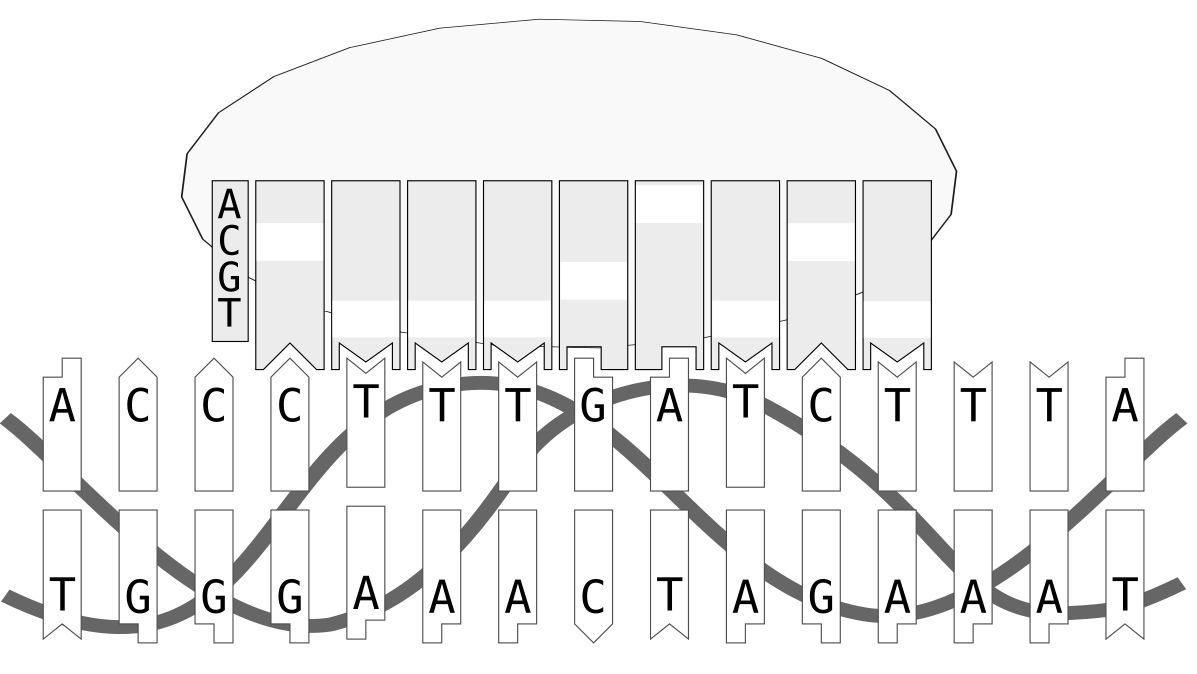
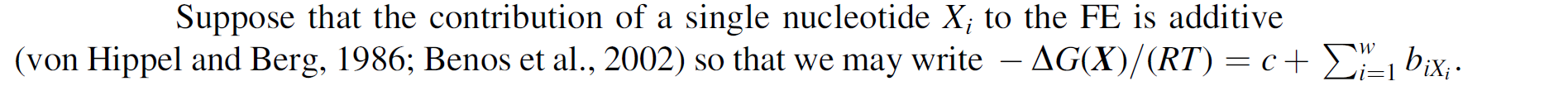
Позиционно-весовая матрица

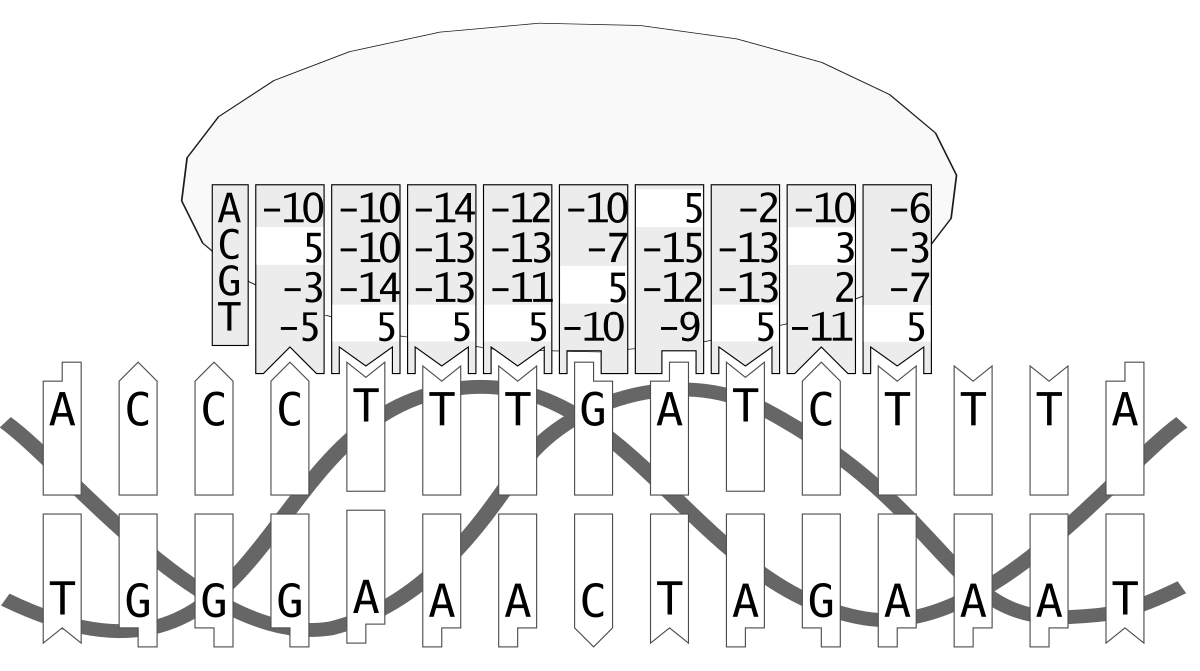
Позиционно-весовая матрица

Stormo, 1982, NAR
Позиционно-весовая матрица
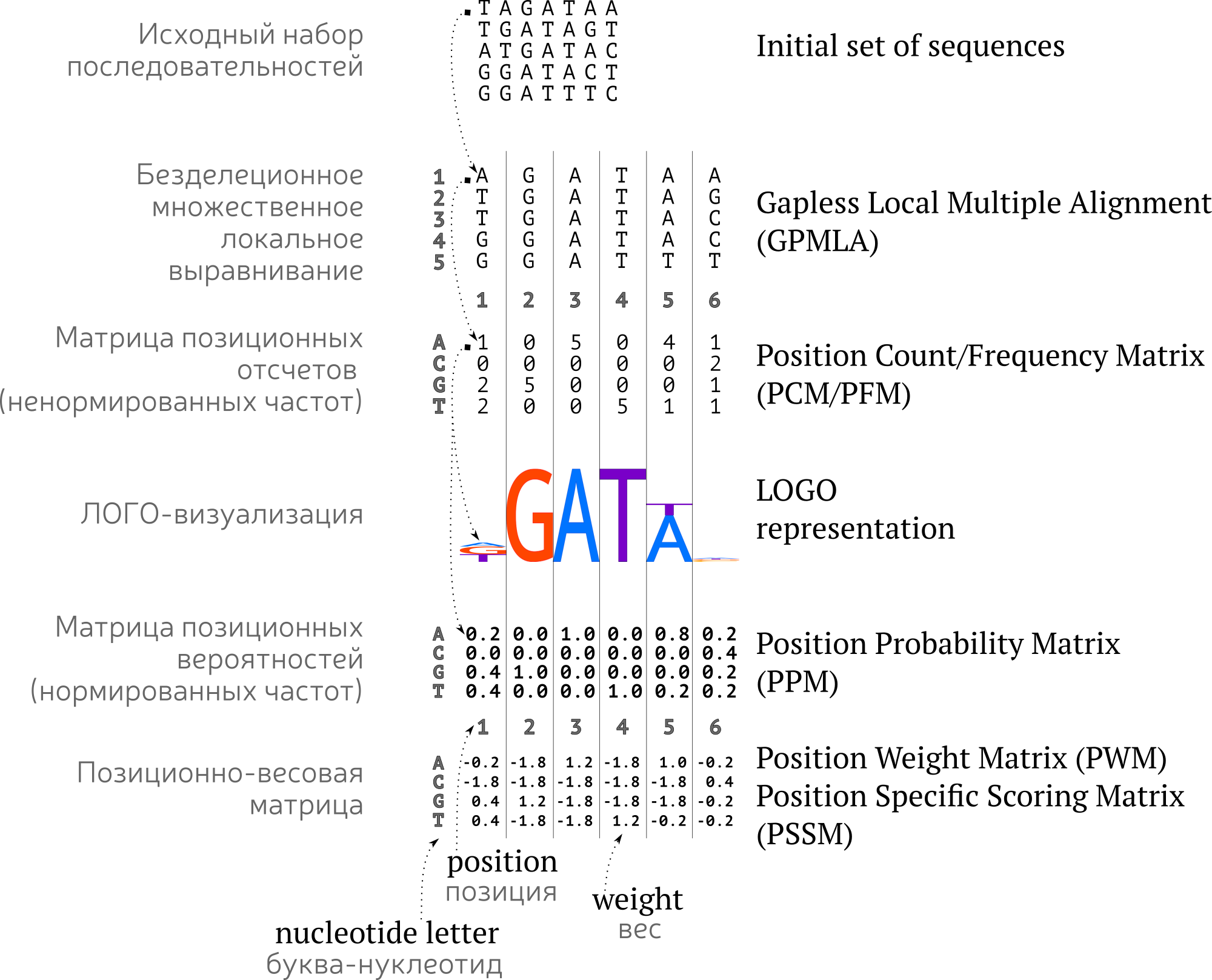
 From a presentation by Gary Stormo
From a presentation by Gary Stormo
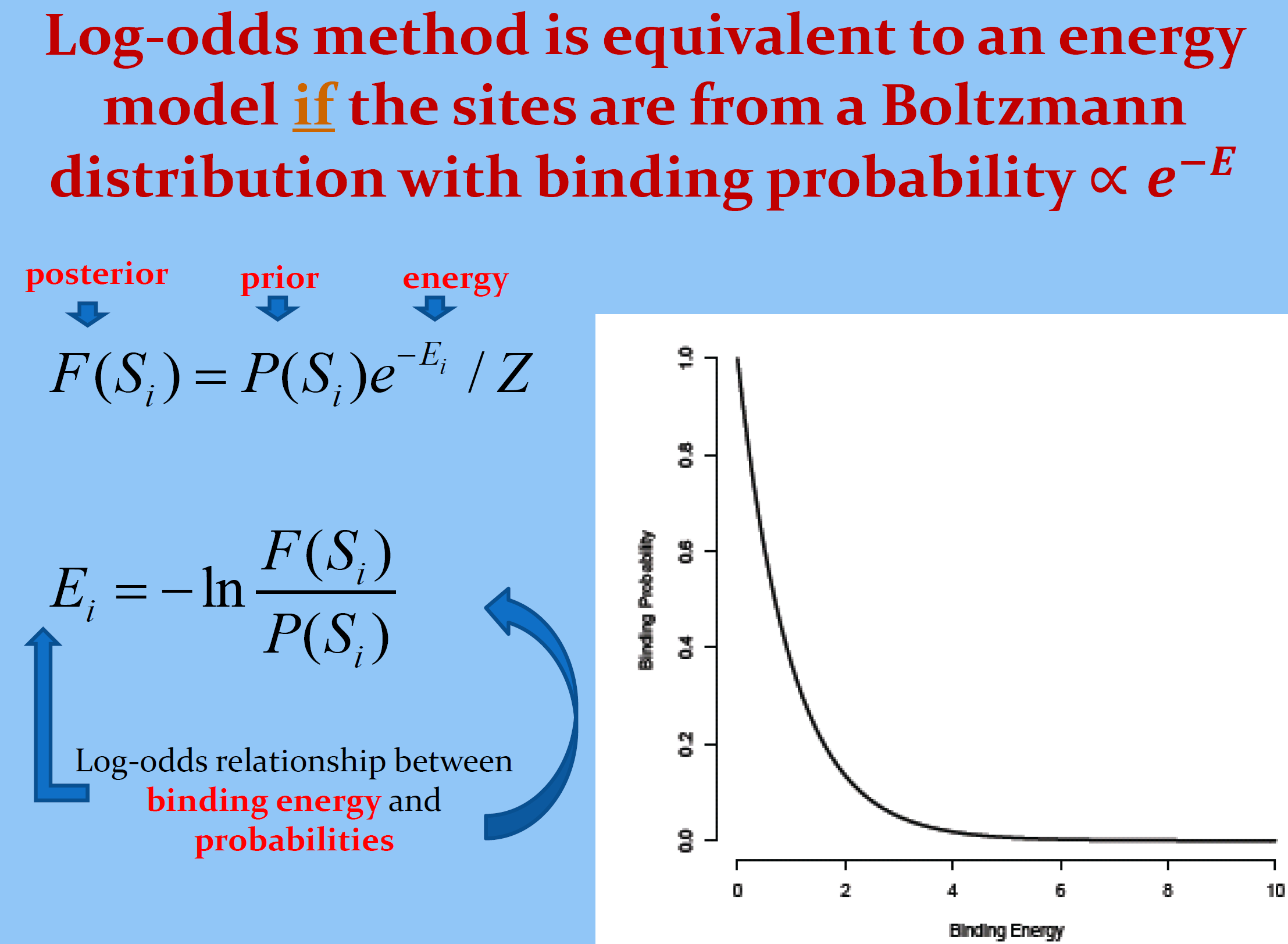
Переход от вероятностей к весам с учетом псевдоотсчетов

Правило следования Лапласа (+1) → Распределение Дирихле
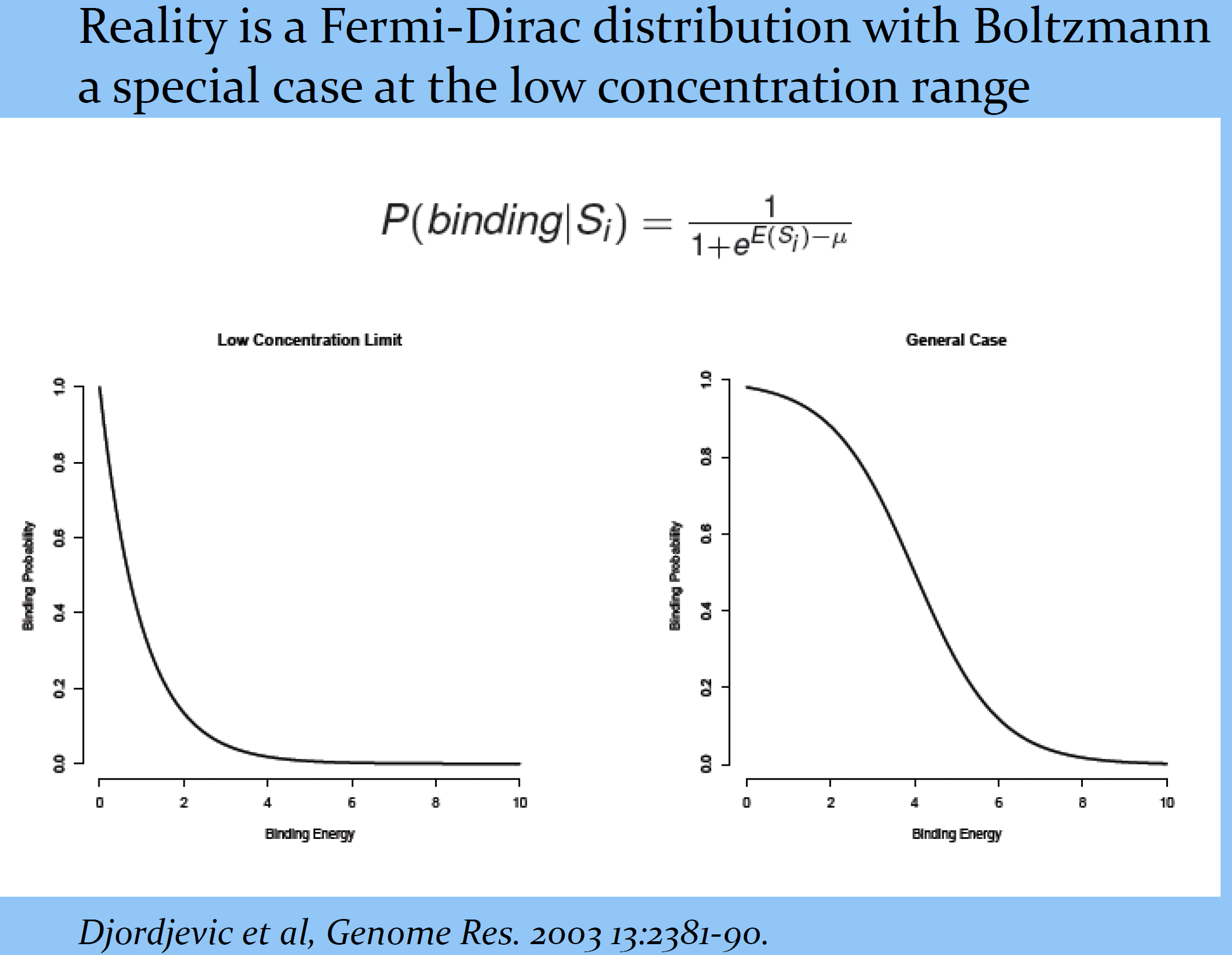 From a presentation by Gary Stormo
From a presentation by Gary Stormo
Информационное содержание и визуализация мотивов
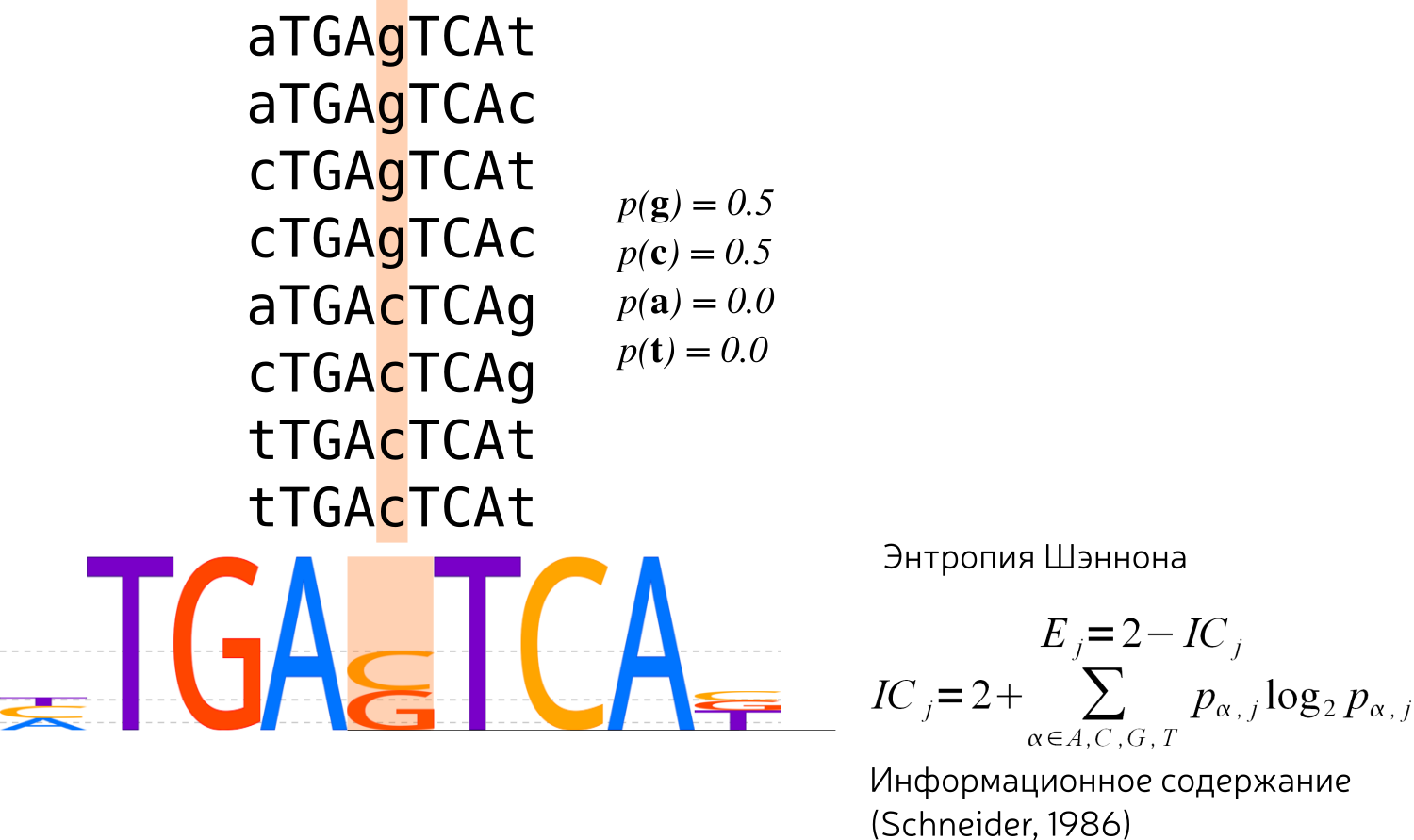
Дискретное информационное содержание
и оптимальность выравнивания
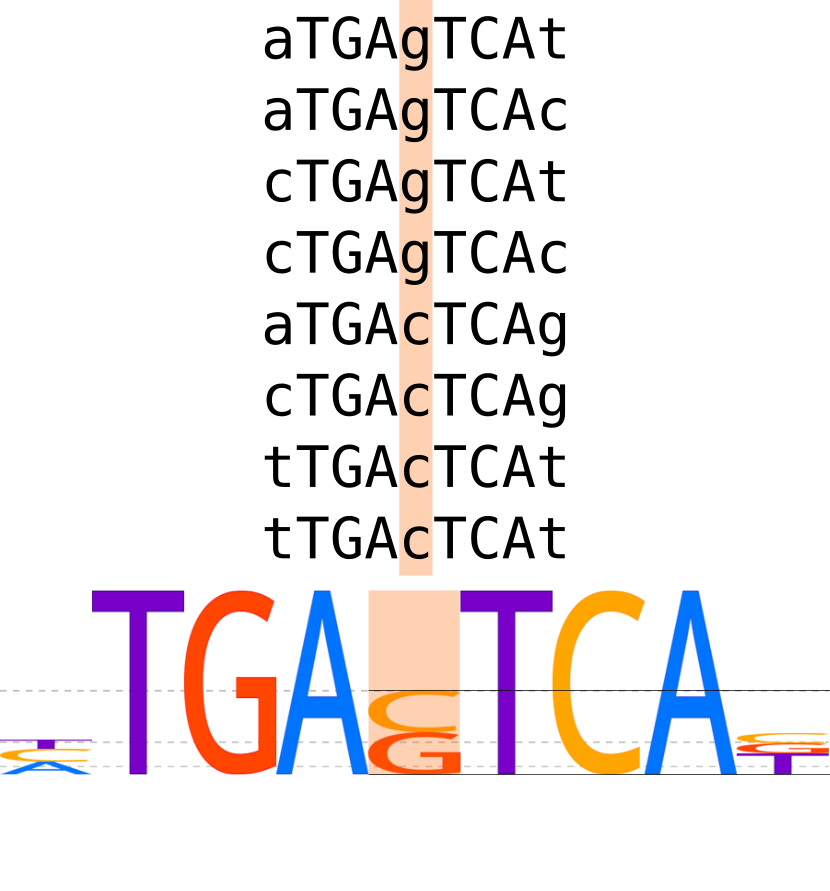
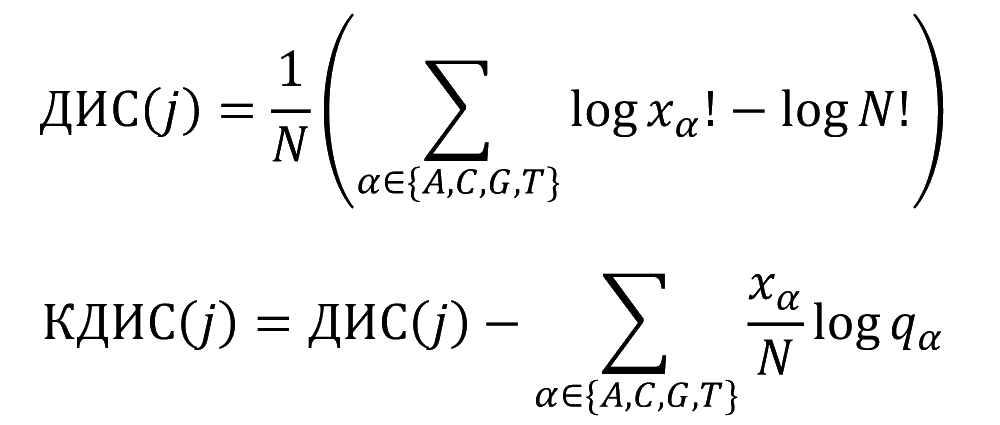
Позиционно-весовая матрица: скоринг слов
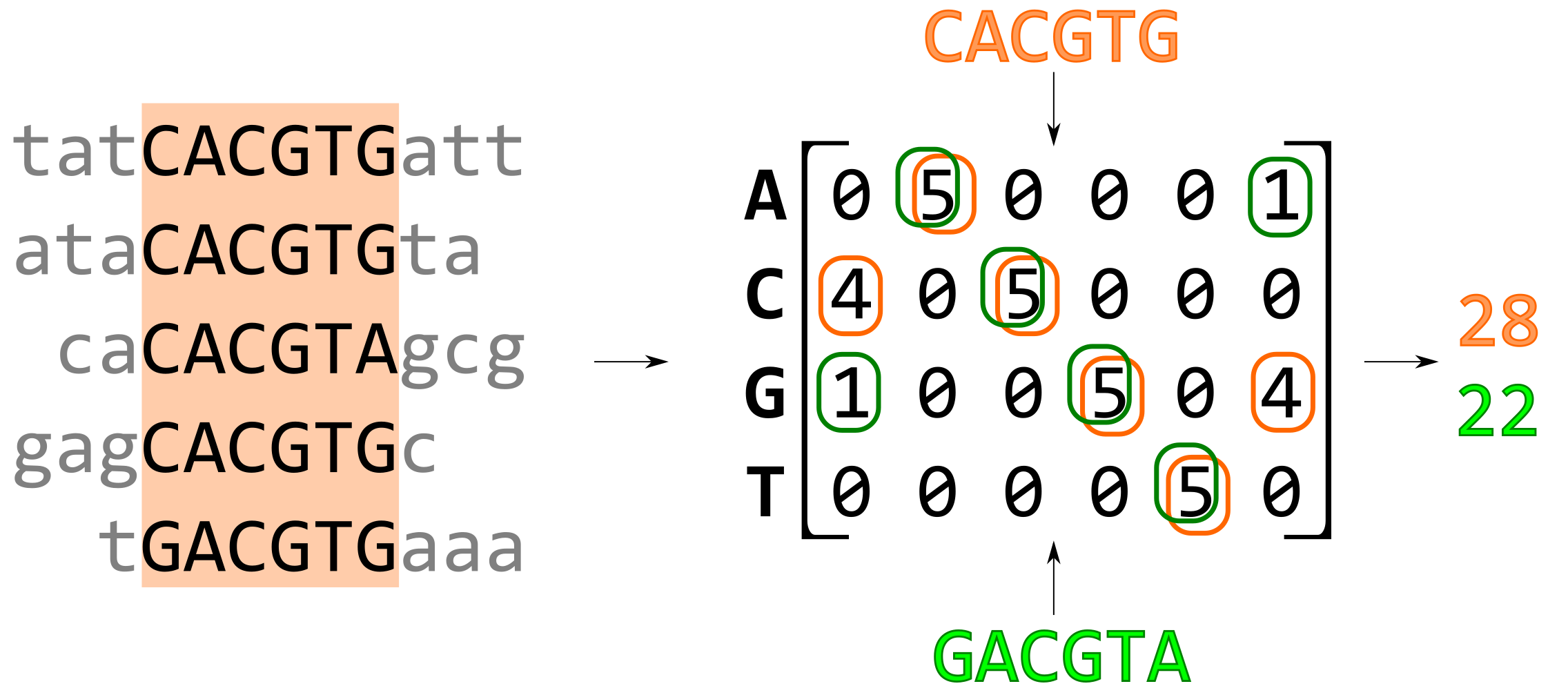
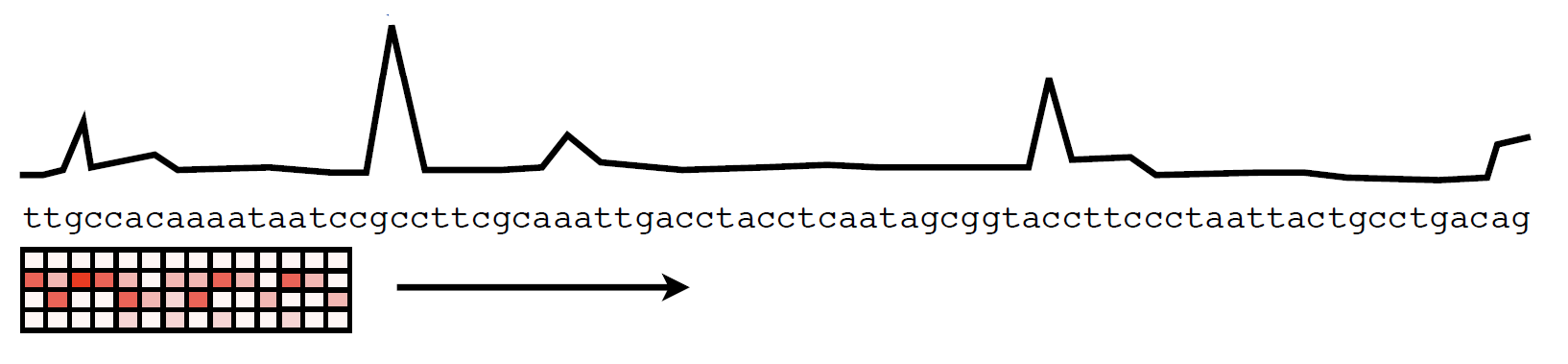
Позиционно-весовая матрица: поиск вхождений
От весовой матрицы обратно к списку слов
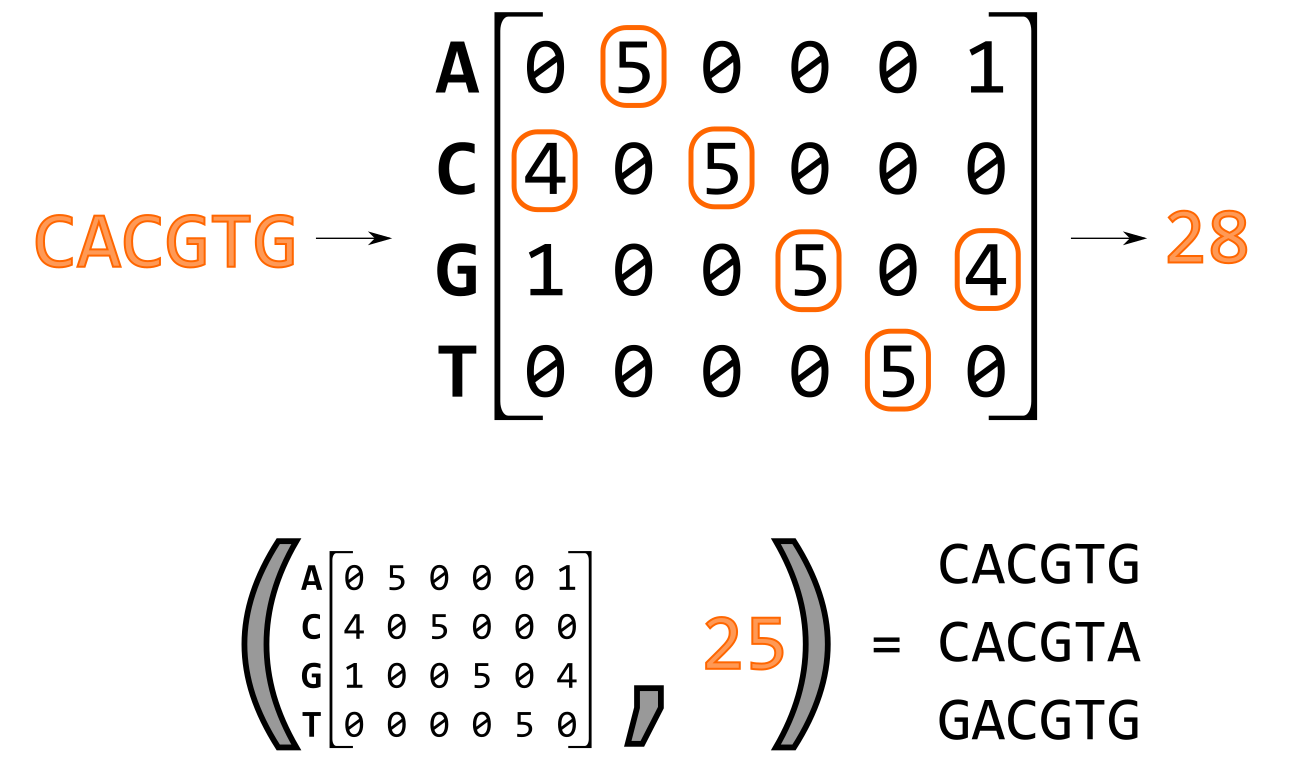
От весовой матрицы обратно к списку слов
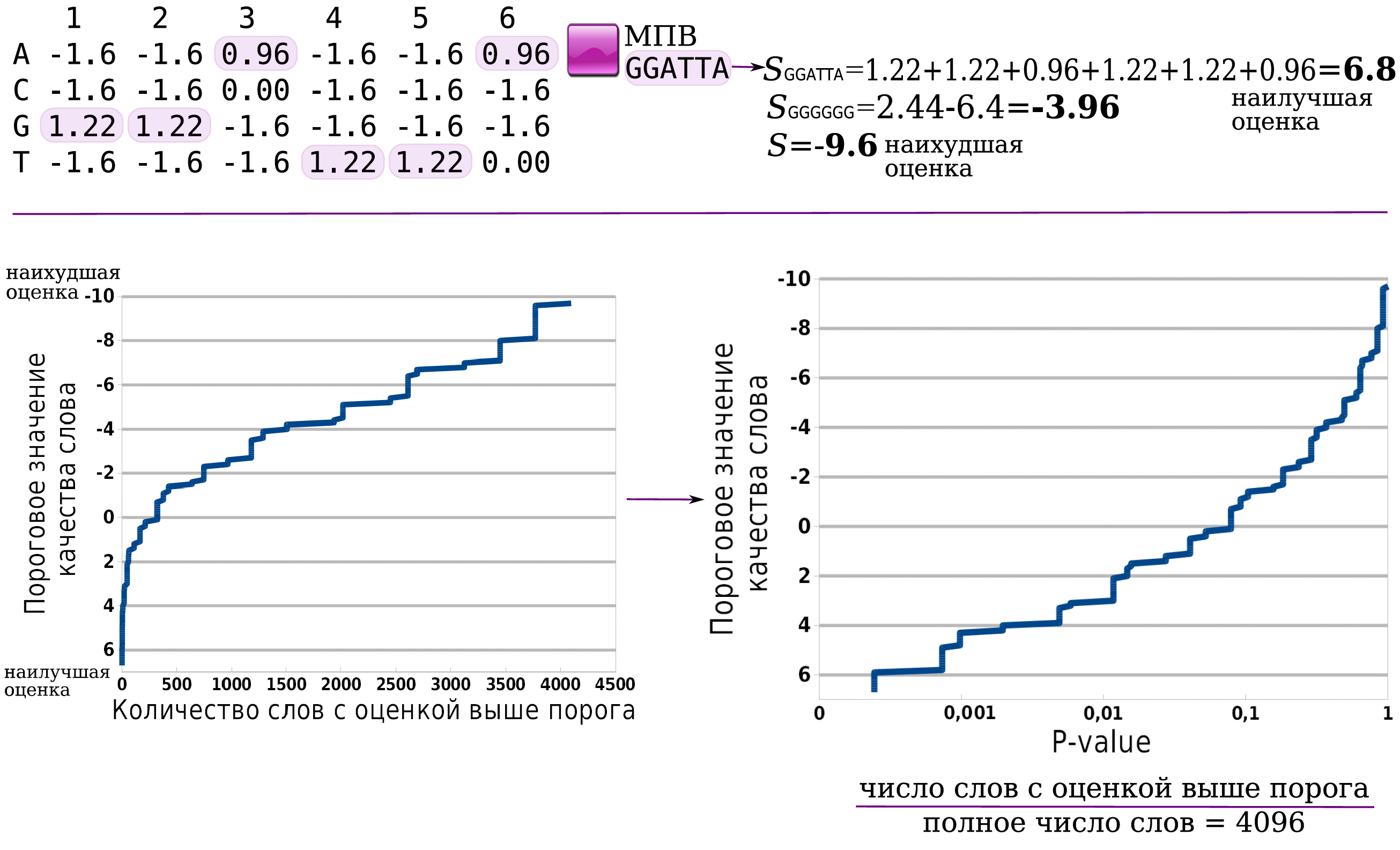
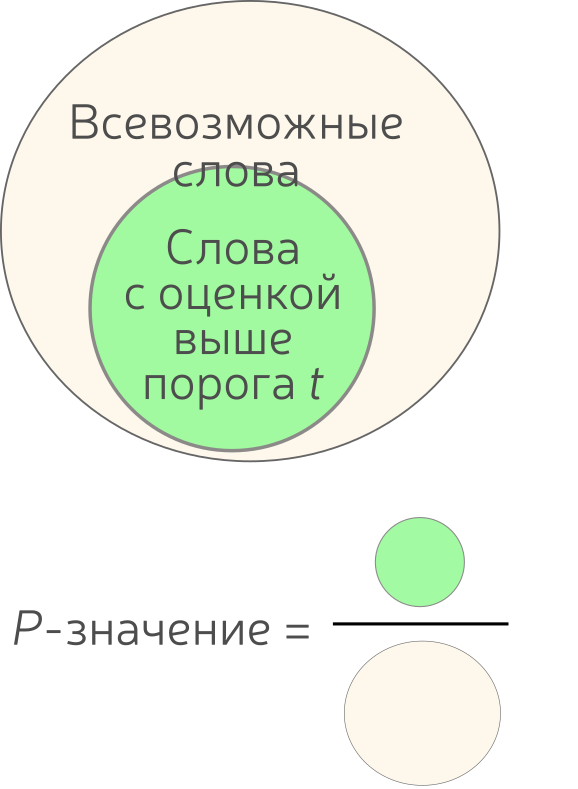
Унификация шкалы с помощью P-значений
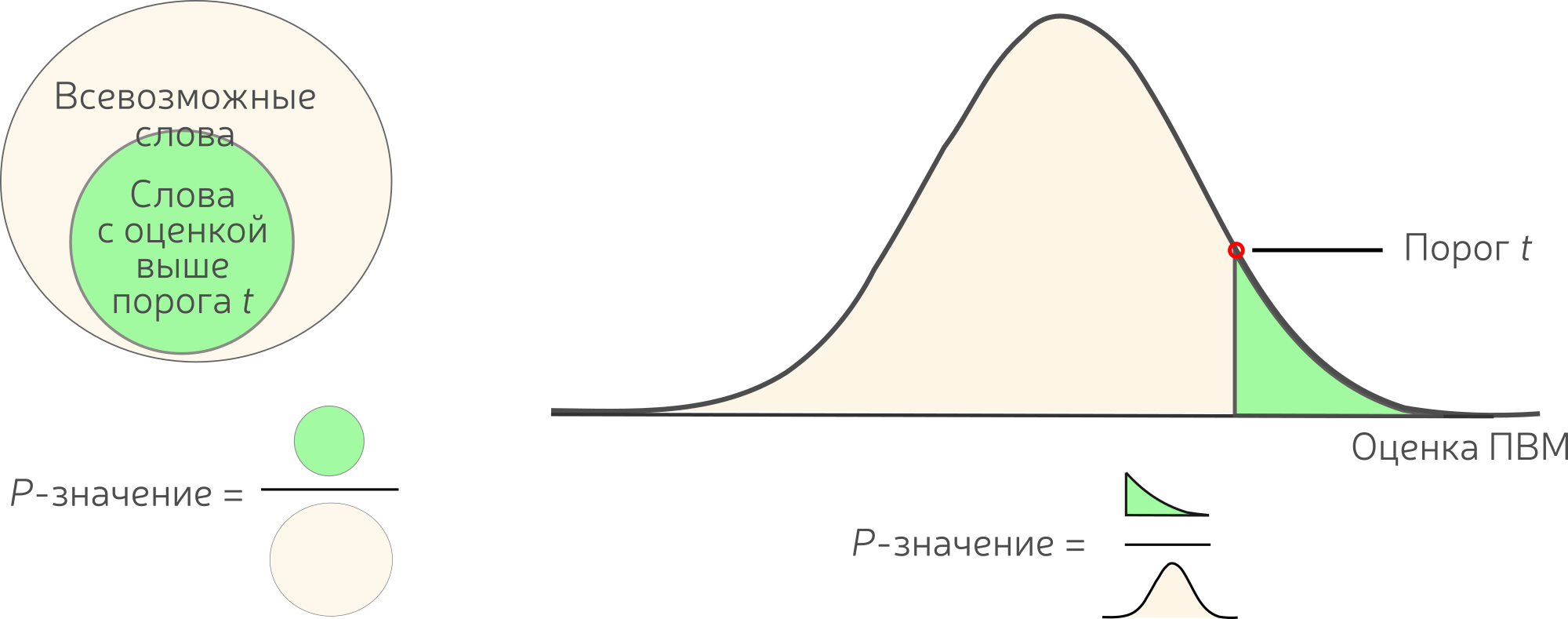
Какие оценки (скоры) хороши?
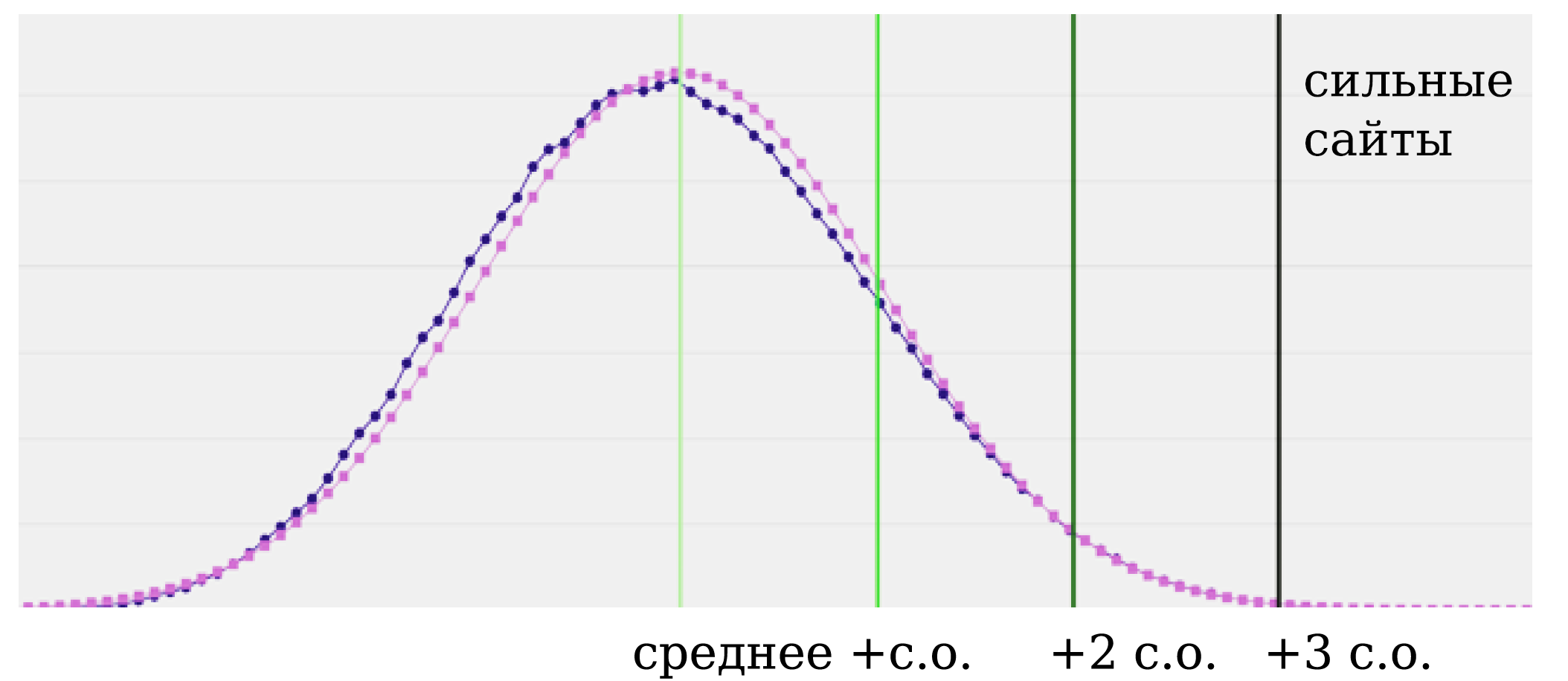
Шеф, у нас снова проблема
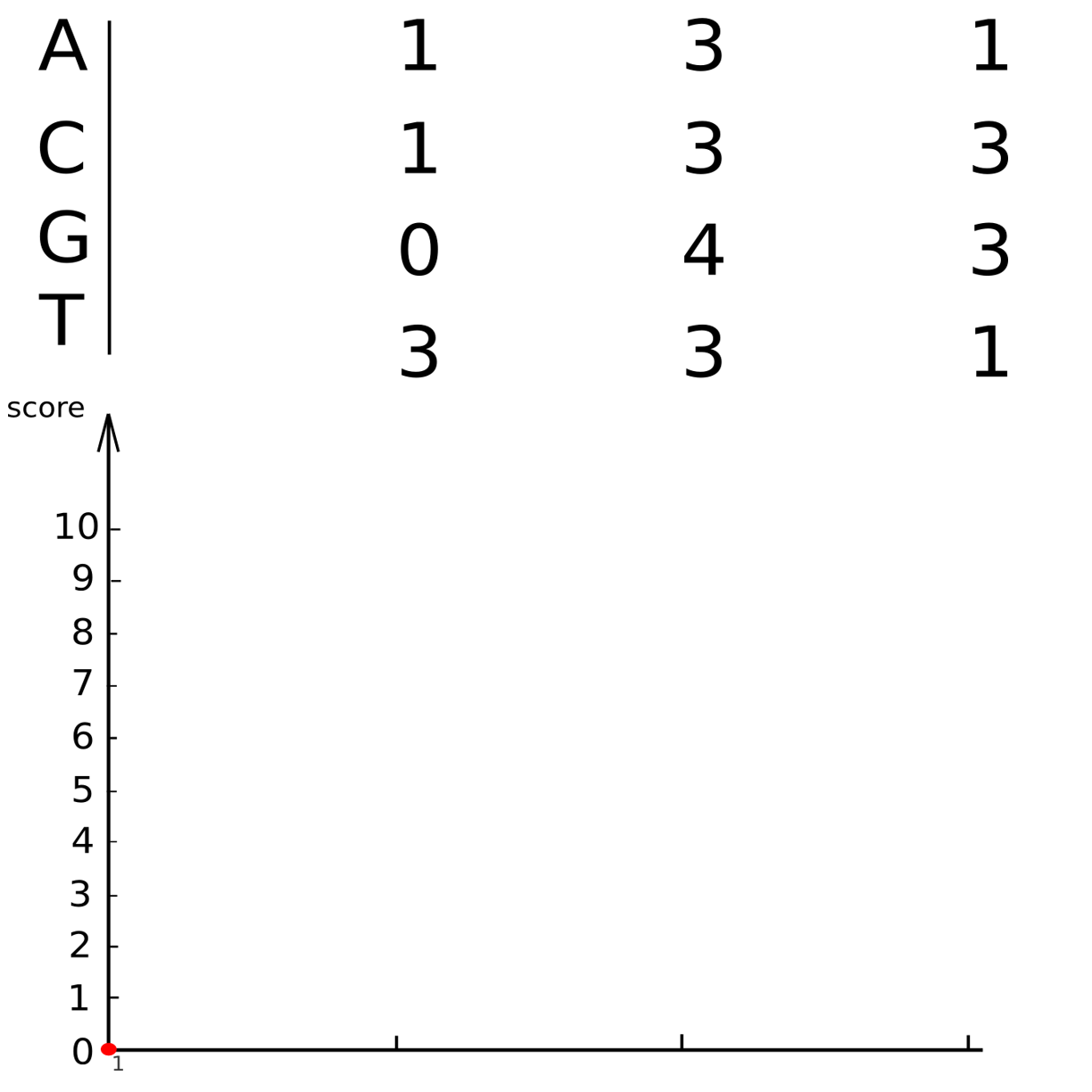
Динамическое программирование
(упрощенный метод Тузе-Варре)
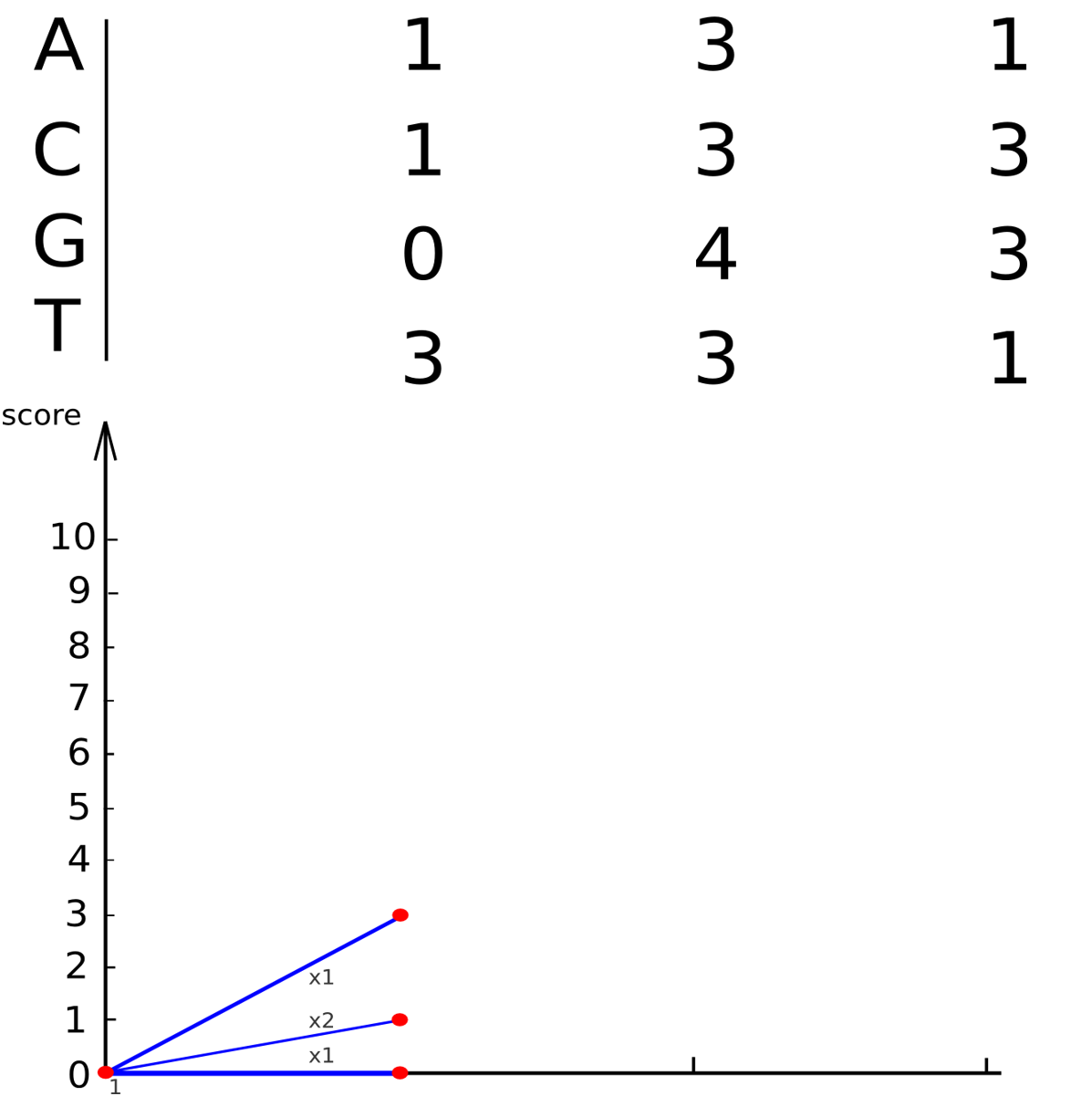
Динамическое программирование
(упрощенный метод Тузе-Варре)
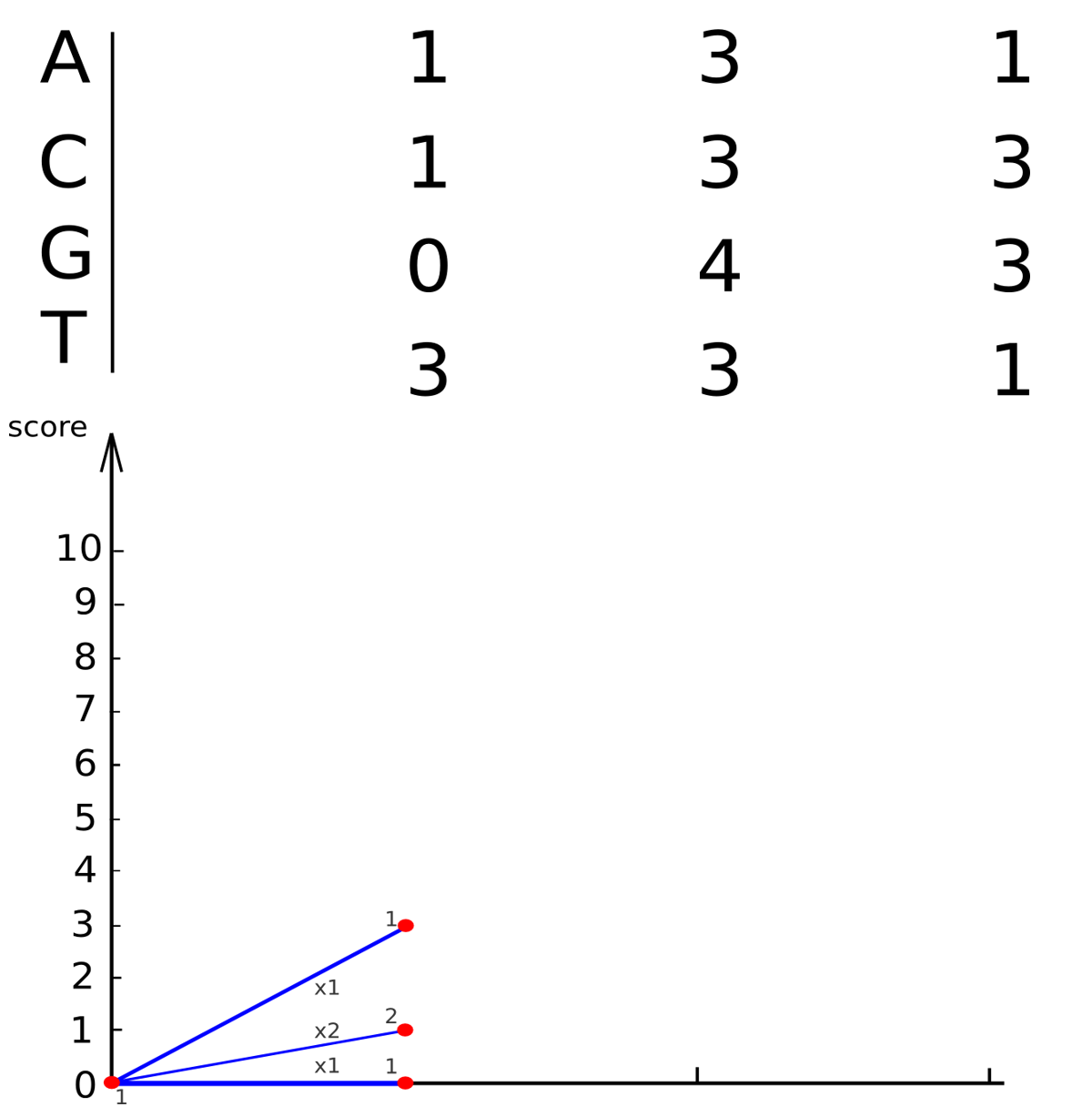
Динамическое программирование
(упрощенный метод Тузе-Варре)
Динамическое программирование
(упрощенный метод Тузе-Варре)
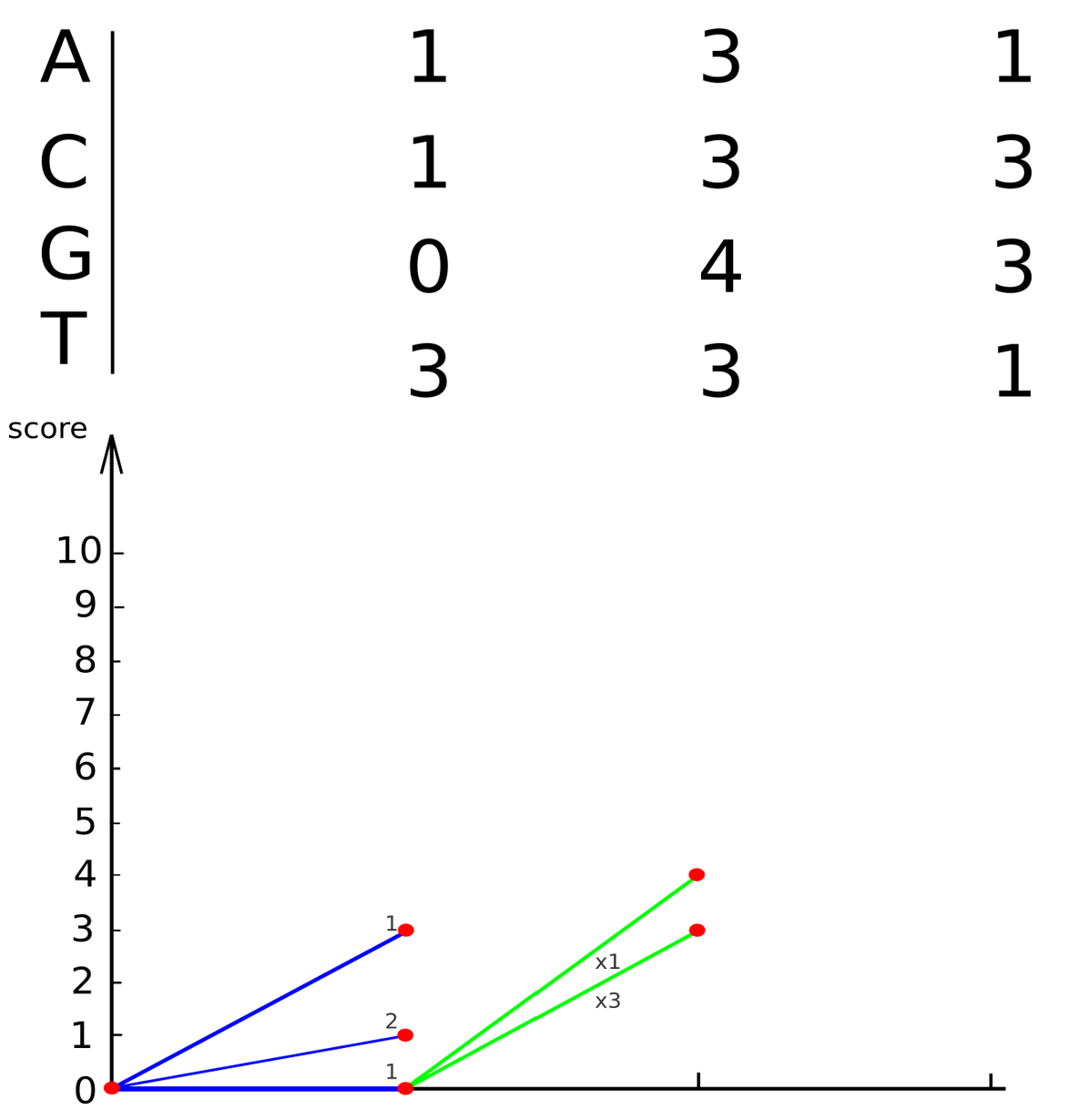
Динамическое программирование
(упрощенный метод Тузе-Варре)
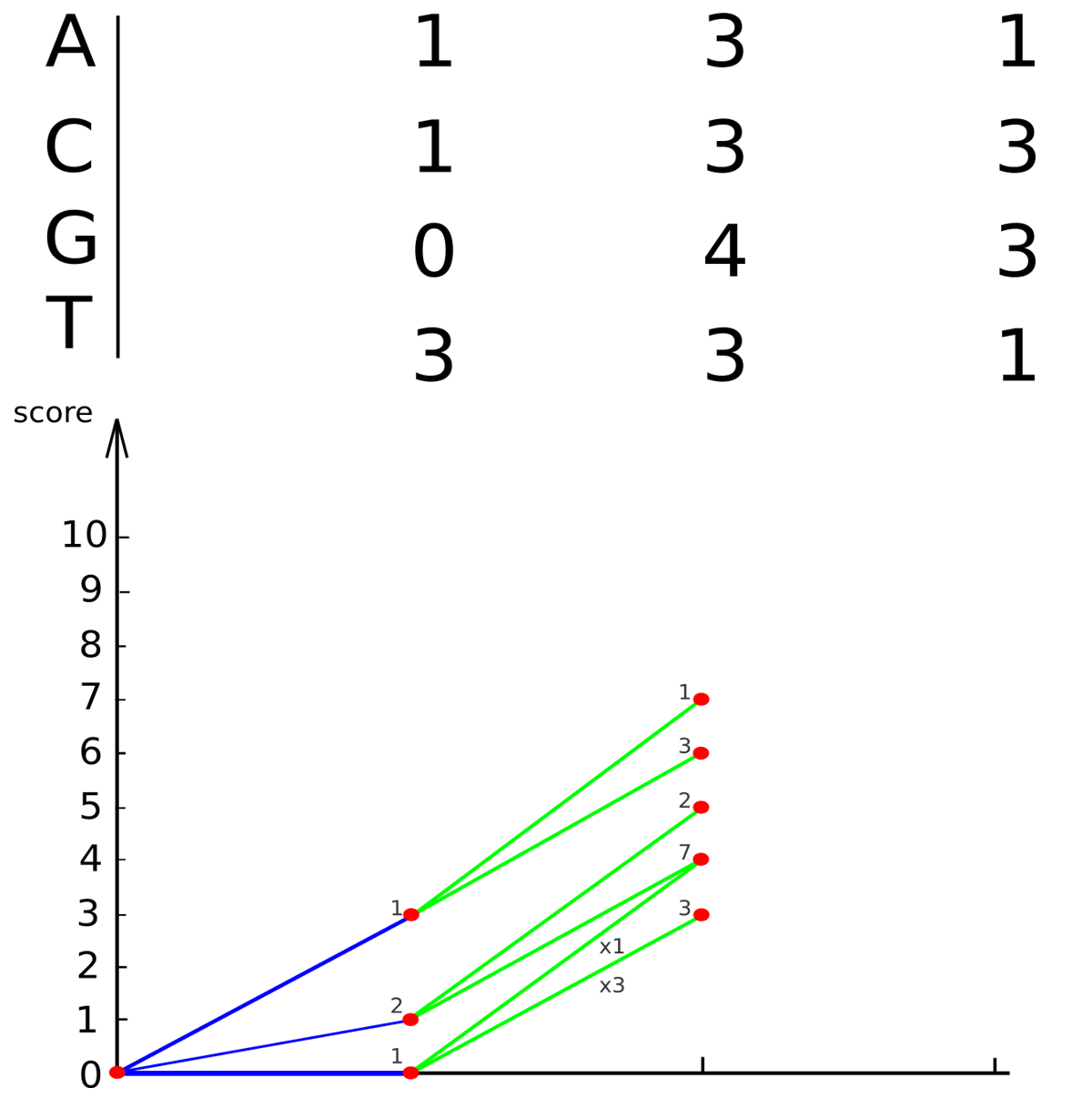
Динамическое программирование
(упрощенный метод Тузе-Варре)
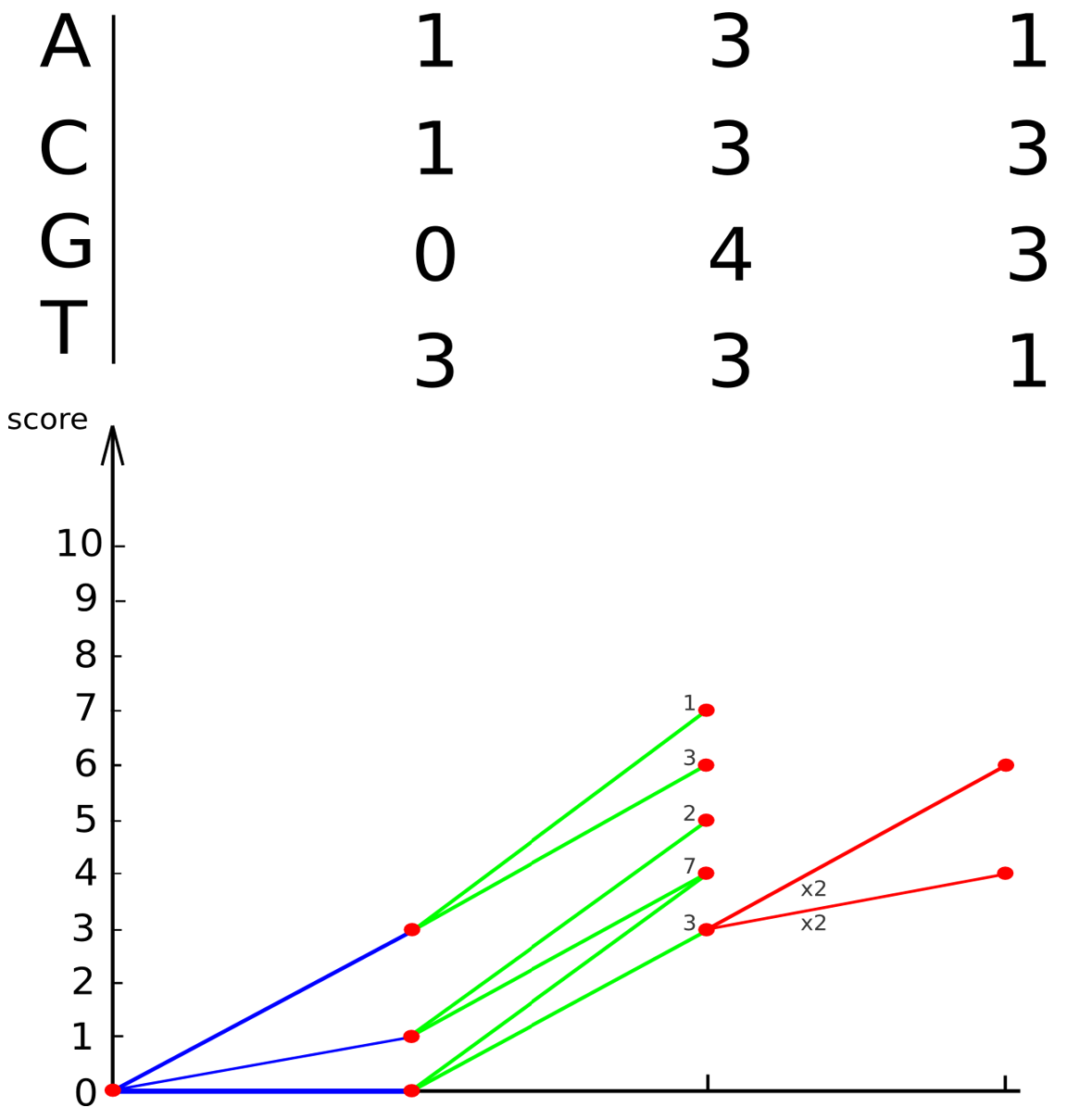
Динамическое программирование
(упрощенный метод Тузе-Варре)
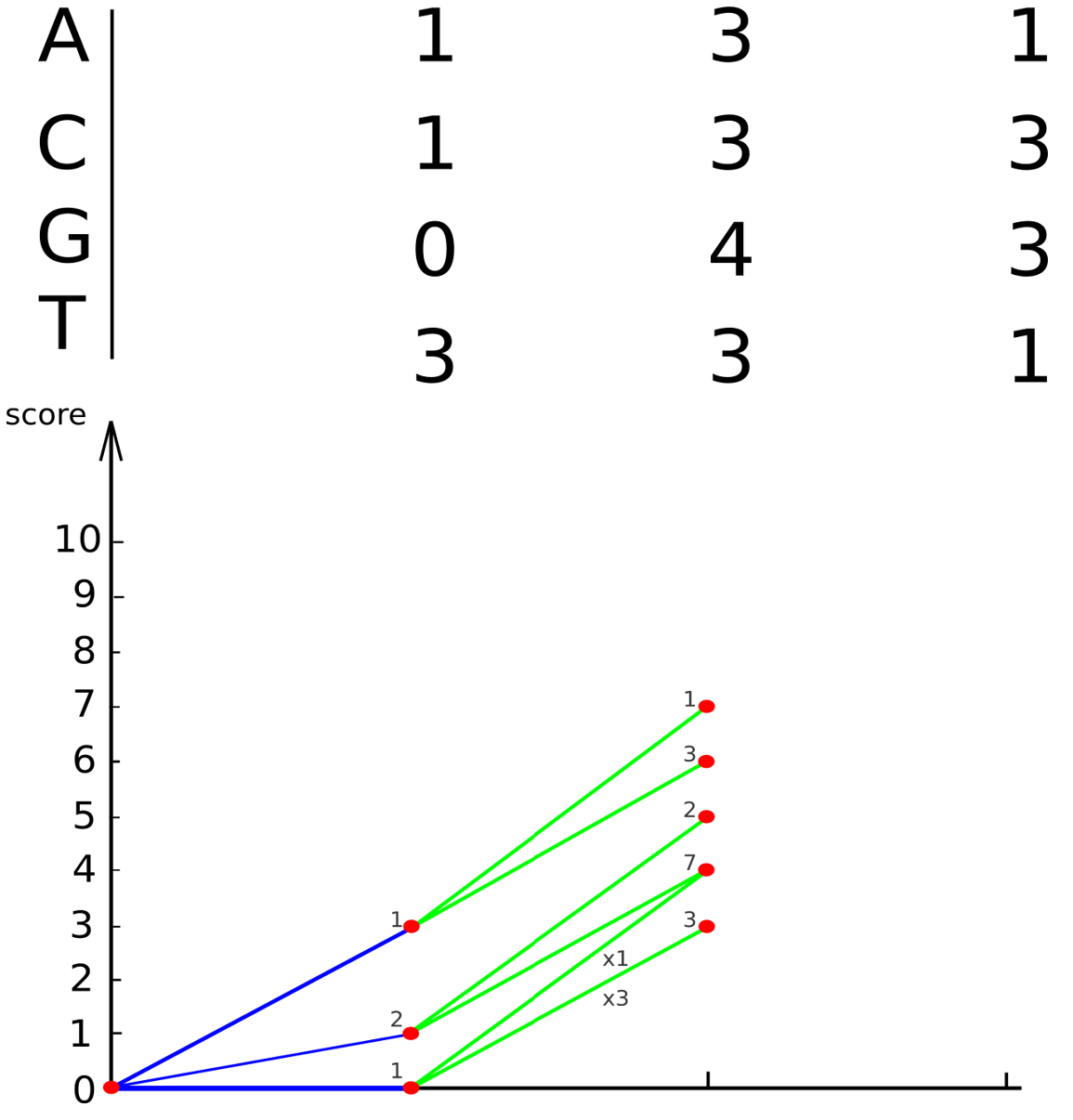
Динамическое программирование
(упрощенный метод Тузе-Варре)
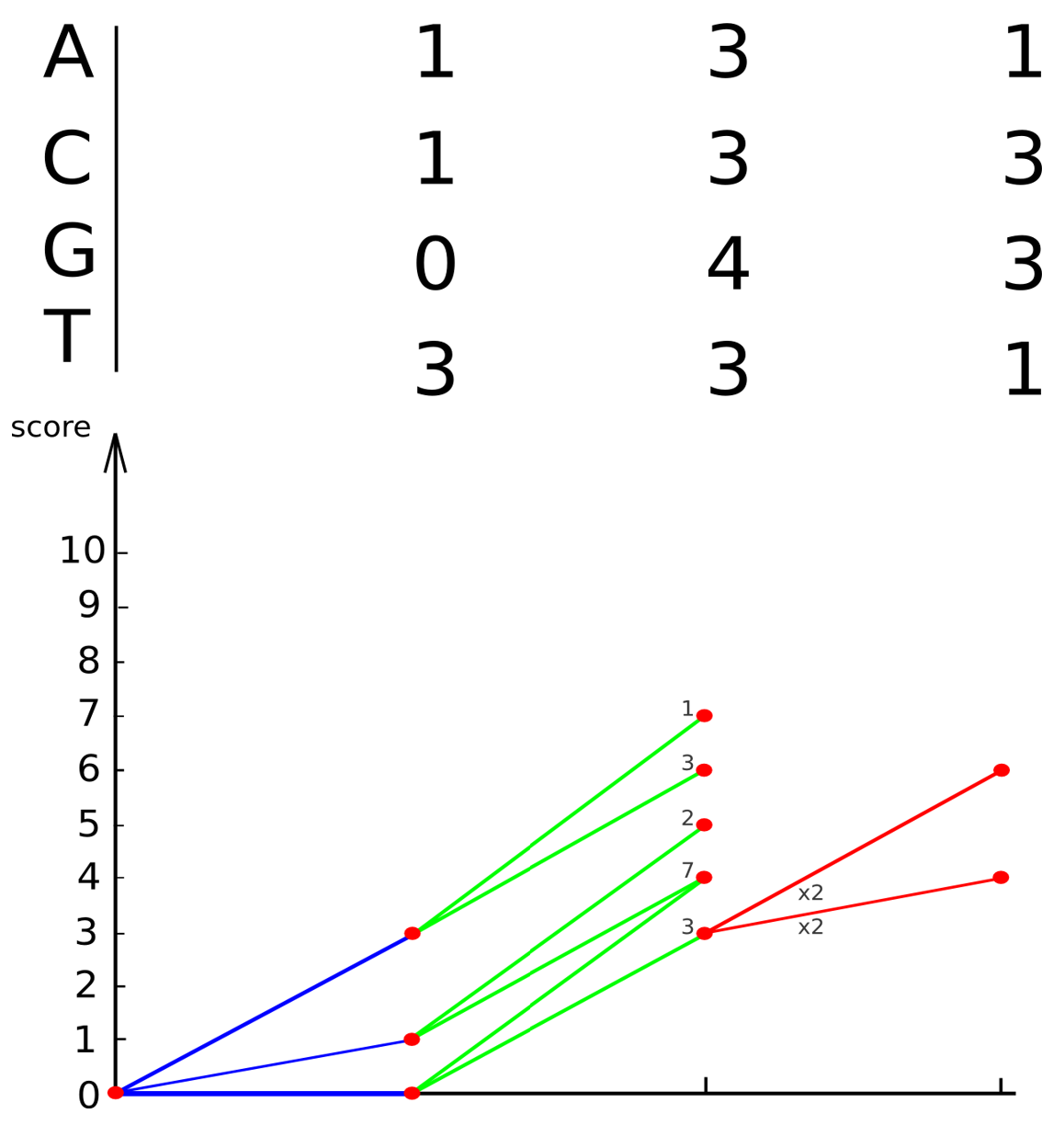
Динамическое программирование
(упрощенный метод Тузе-Варре)
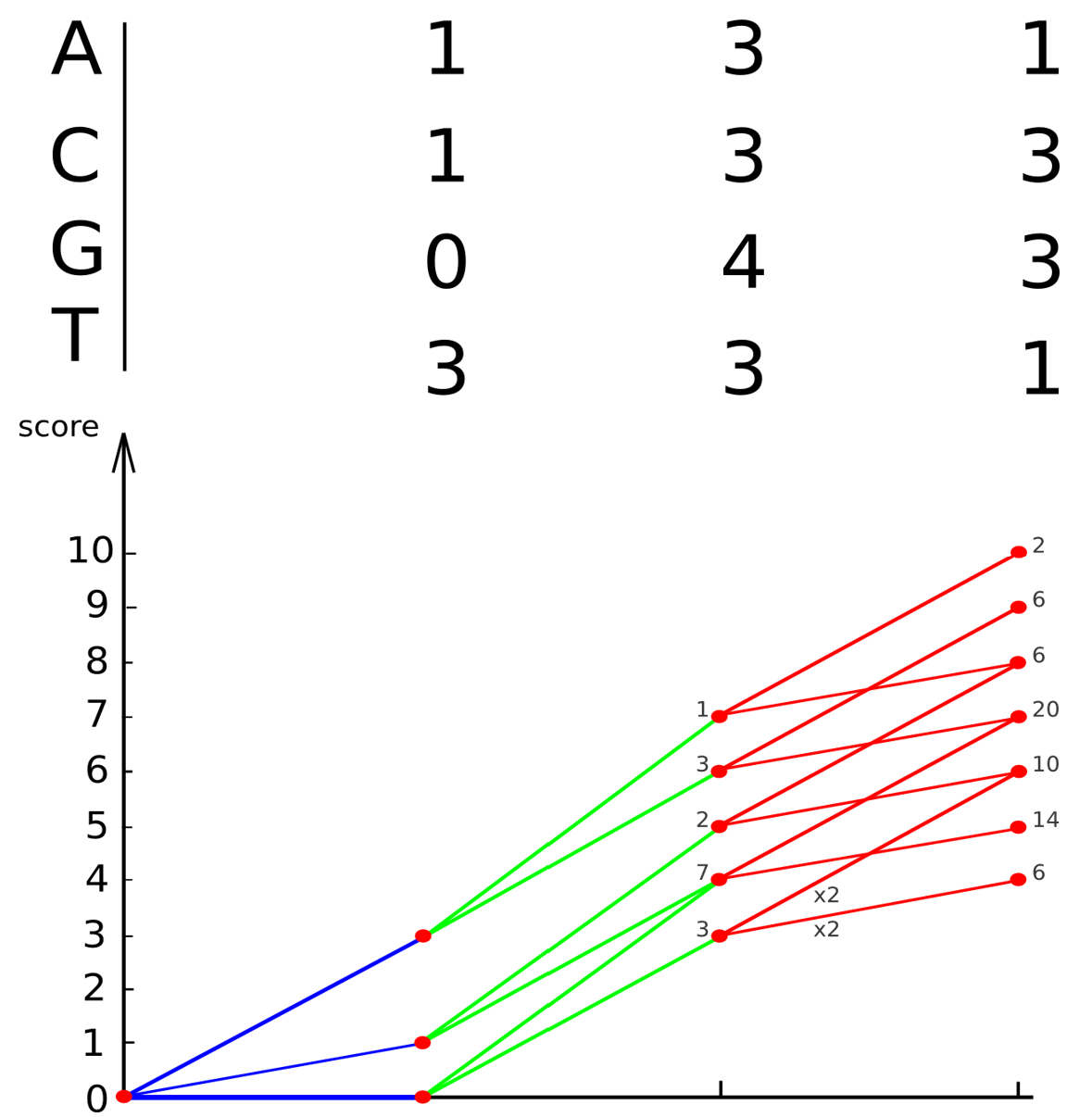
Динамическое программирование
(упрощенный метод Тузе-Варре)
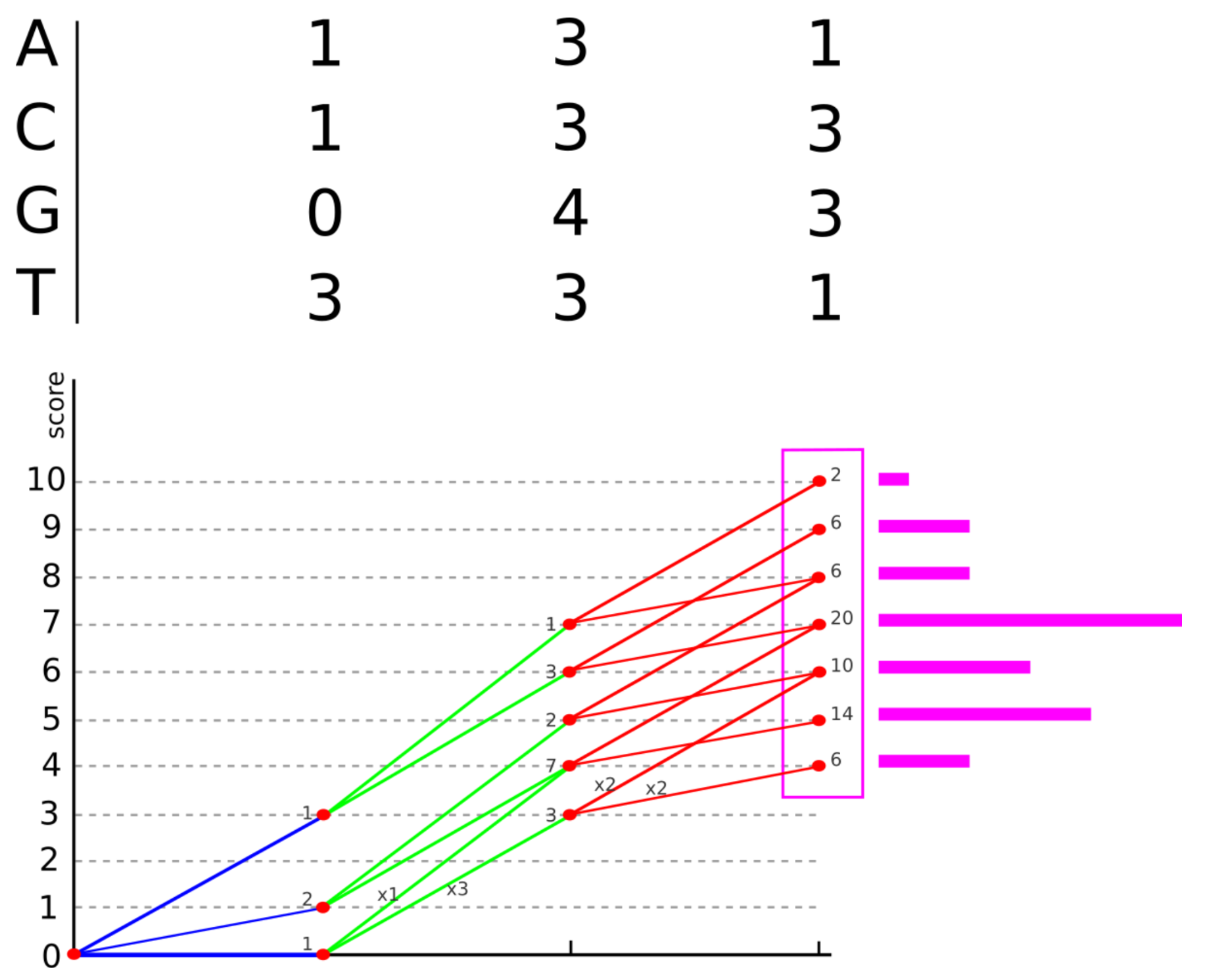
Позиционно-весовая матрица: поиск вхождений
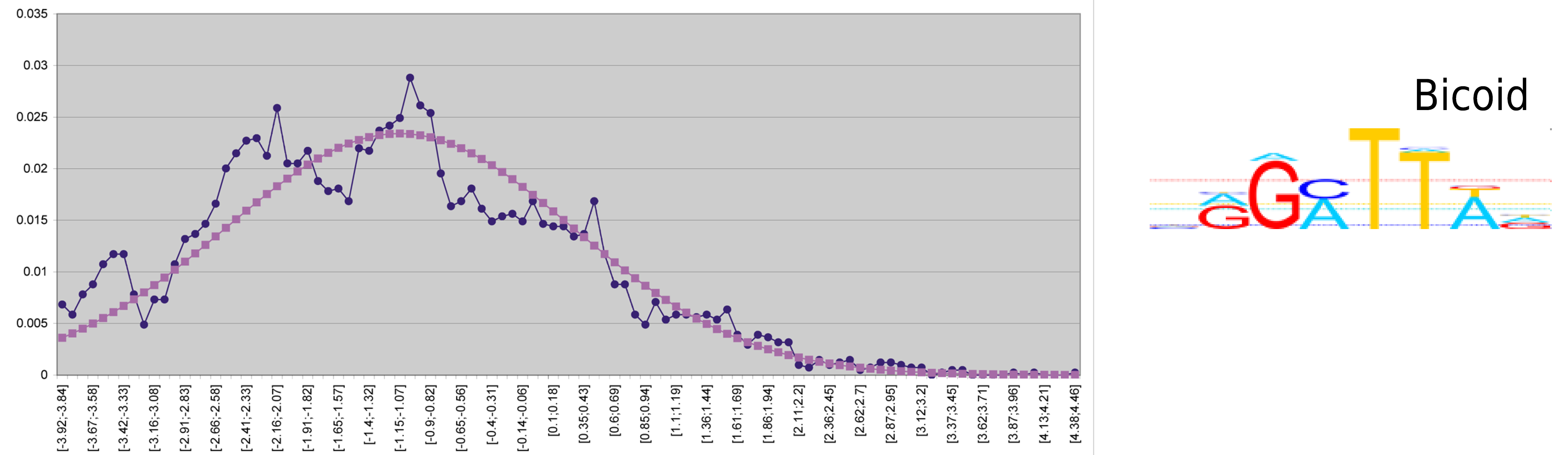
предсказания достоверны для коротких олигонуклеотидов

для последовательности длины 1000


Модели и методы идентификации мотива
Список слов, консенсус, регулярное выражение ⇠ дискретные (словарные) методы⇩
Позиционно-весовая матрица ⇠ EM-алгоритм, сэмплирование по Гиббсу, жадная оптимизация
⇩
Сложная модель ⇠ машинное обучение
Любимая задача биоинформатиков
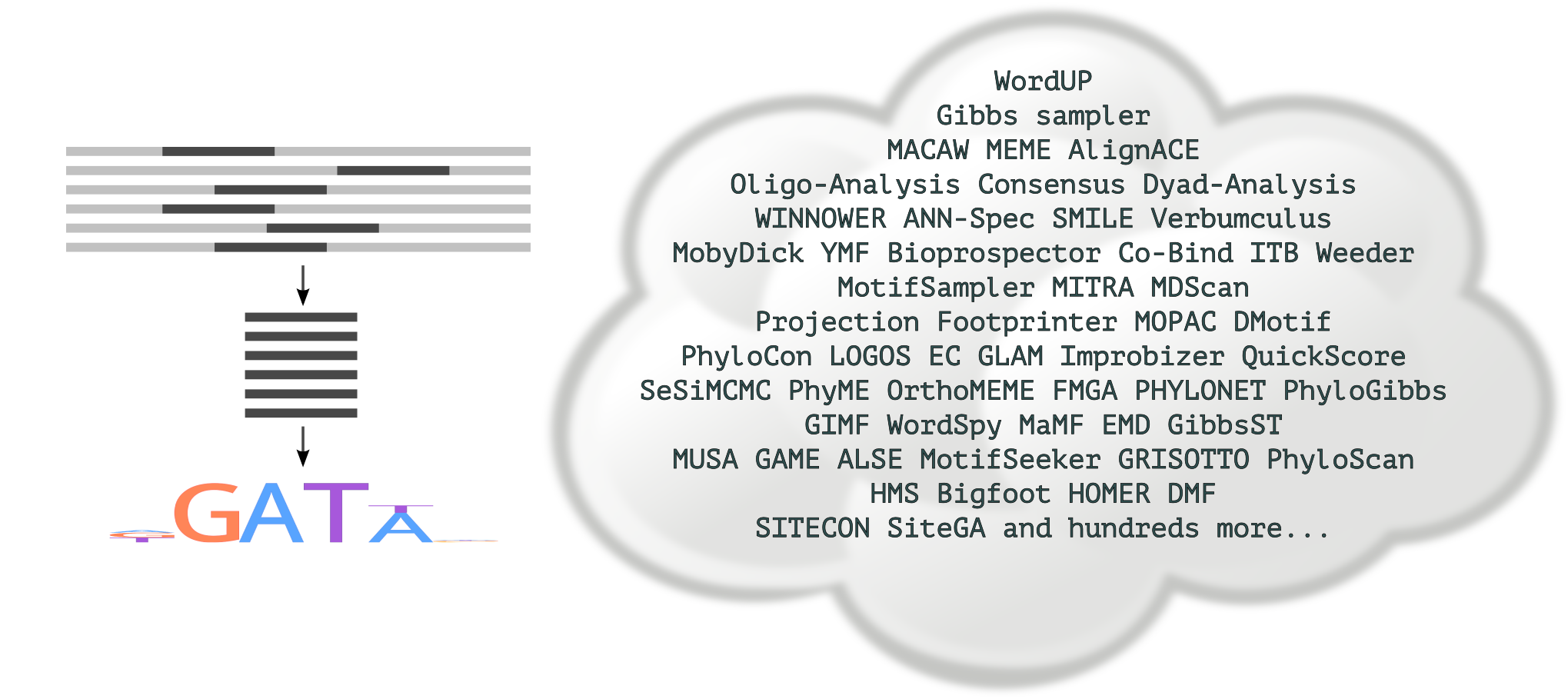 Wei et al. 2007 - 120 software tools, Quest et al. 2008 - 150 software tools
Wei et al. 2007 - 120 software tools, Quest et al. 2008 - 150 software tools
Задача не поставлена, решение не однозначно





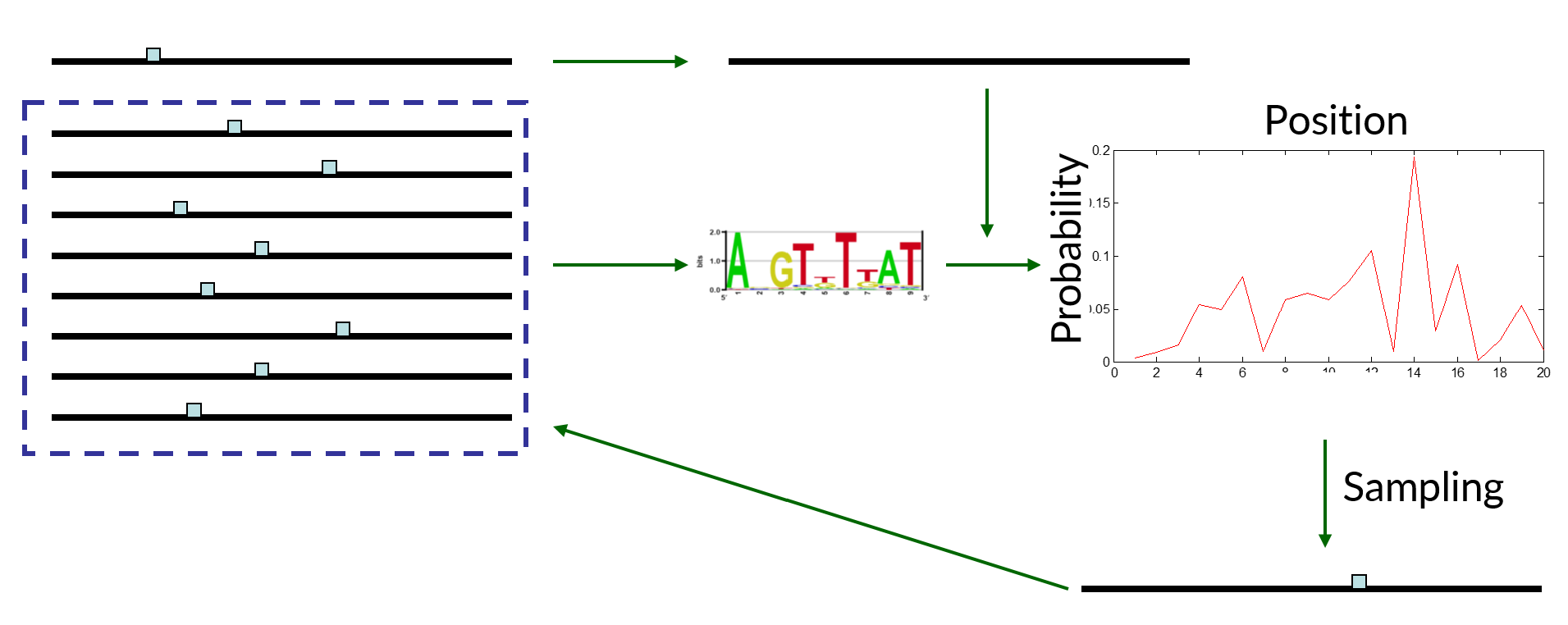
EM-алгоритм (Expectation-Maximization)
 Jianhua Ruan, http://www.cs.utsa.edu/~jruan/teaching/cs5263_spring_2017/index.htm
Jianhua Ruan, http://www.cs.utsa.edu/~jruan/teaching/cs5263_spring_2017/index.htm
EM-алгоритм (Expectation-Maximization)
 Jianhua Ruan, http://www.cs.utsa.edu/~jruan/teaching/cs5263_spring_2017/index.htm
Jianhua Ruan, http://www.cs.utsa.edu/~jruan/teaching/cs5263_spring_2017/index.htm
EM-алгоритм (Expectation-Maximization)
 Jianhua Ruan, http://www.cs.utsa.edu/~jruan/teaching/cs5263_spring_2017/index.htm
Jianhua Ruan, http://www.cs.utsa.edu/~jruan/teaching/cs5263_spring_2017/index.htm
EM-алгоритм (Expectation-Maximization)
 Jianhua Ruan, http://www.cs.utsa.edu/~jruan/teaching/cs5263_spring_2017/index.htm
Jianhua Ruan, http://www.cs.utsa.edu/~jruan/teaching/cs5263_spring_2017/index.htm
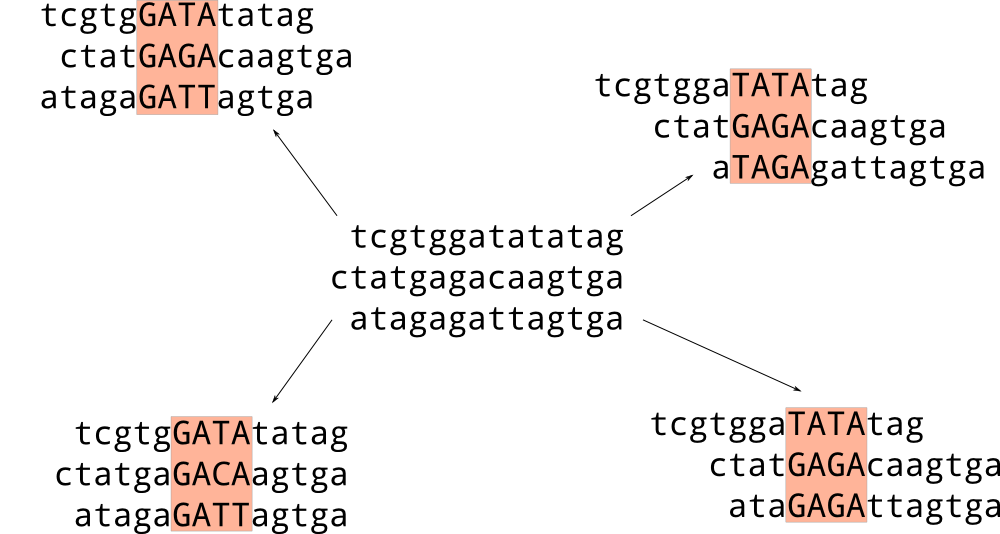
Гиббсовский сэмплер
 Jianhua Ruan, http://www.cs.utsa.edu/~jruan/teaching/cs5263_spring_2017/index.htm
Jianhua Ruan, http://www.cs.utsa.edu/~jruan/teaching/cs5263_spring_2017/index.htm
Гиббсовский сэмплер

The Gruffalo Child

Julia Donaldson, Alex Scheffer

The Gruffalo said
that no gruffalo should
Ever set foot
in the deep dark wood.
"Why not? Why not?"
"Because if you do
The Big Bad Mouse
Data
will be after you."

"What does he look like? Tell me, Dad.
Is he it terribly
BIG and terribly BAD?"
Переход от догеномных к геномным методам

Идентификация мотивов
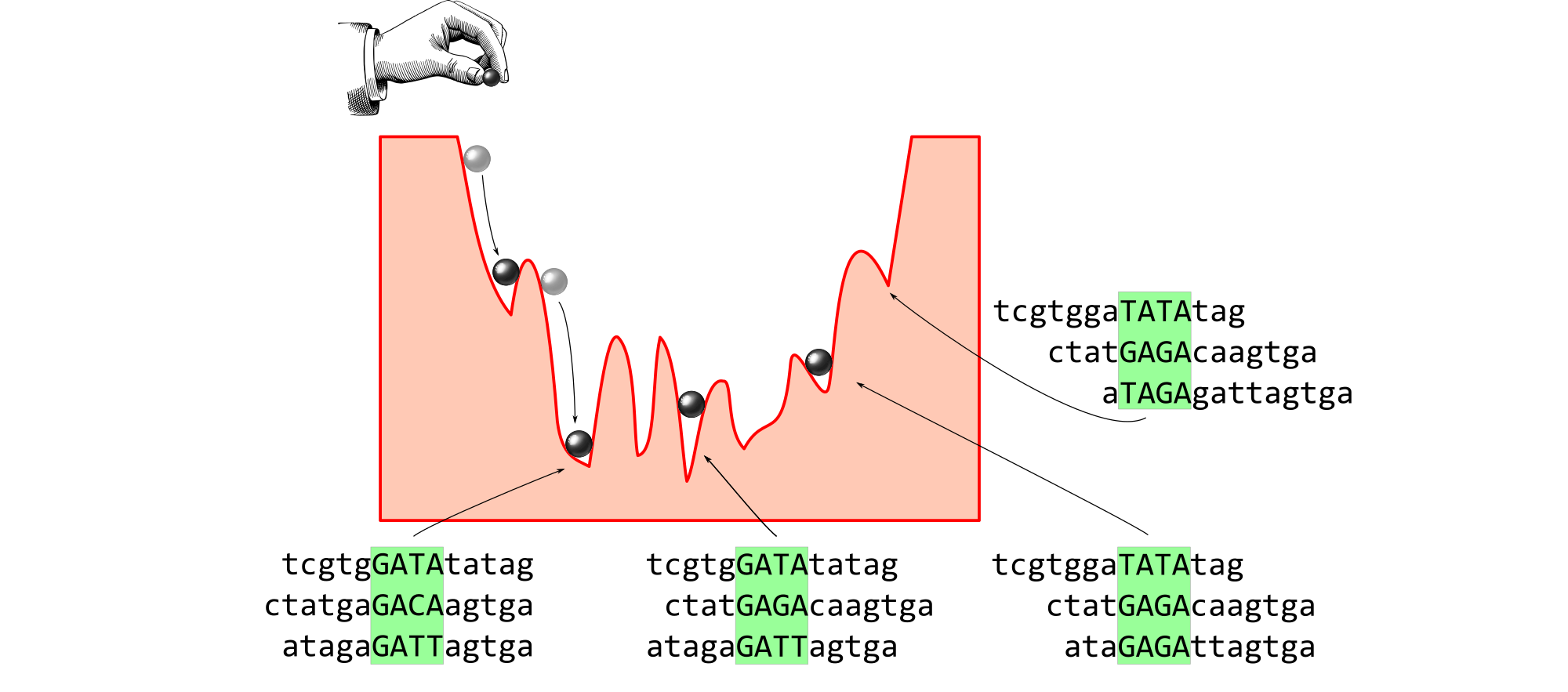
Идентификация мотивов
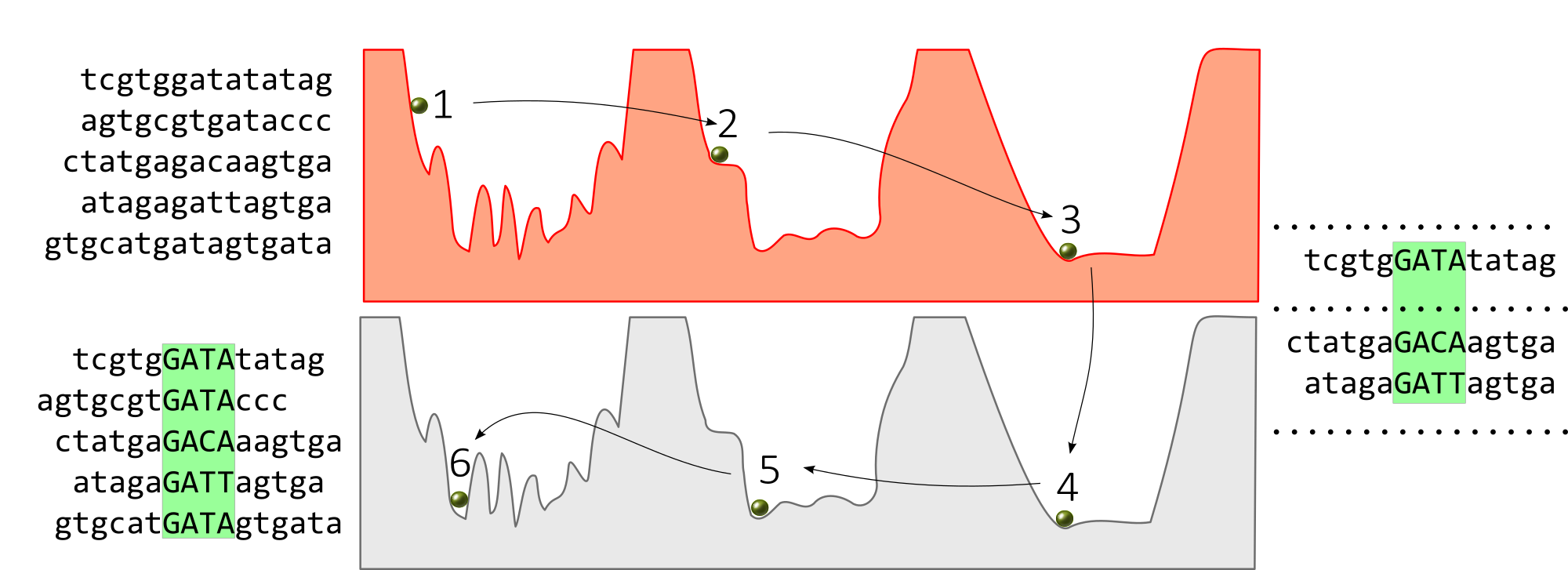
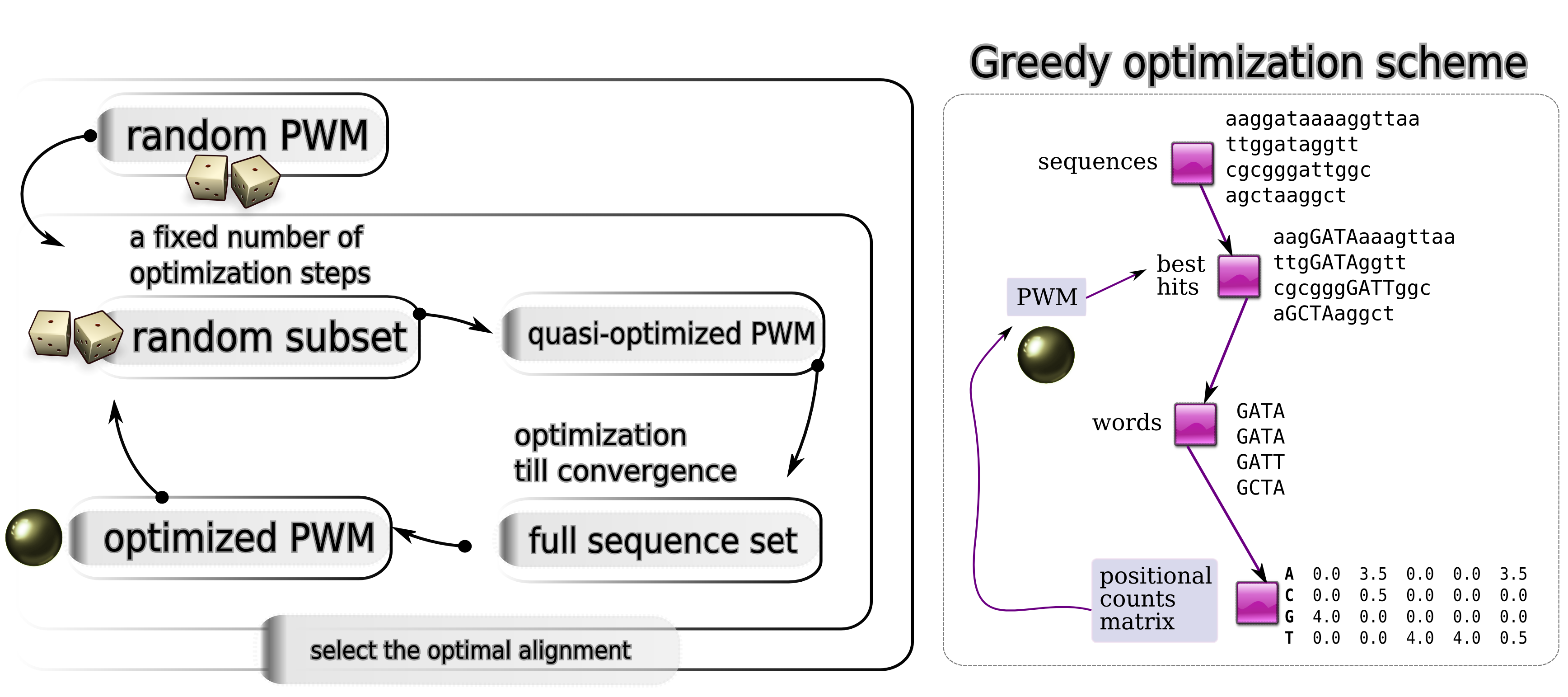
Жадная оптимизация




Идентичны ли два мотива?


Мера Жаккара как "естественная" оценка сходства мотивов
Двухмерный вариант метода Тузе-Варре
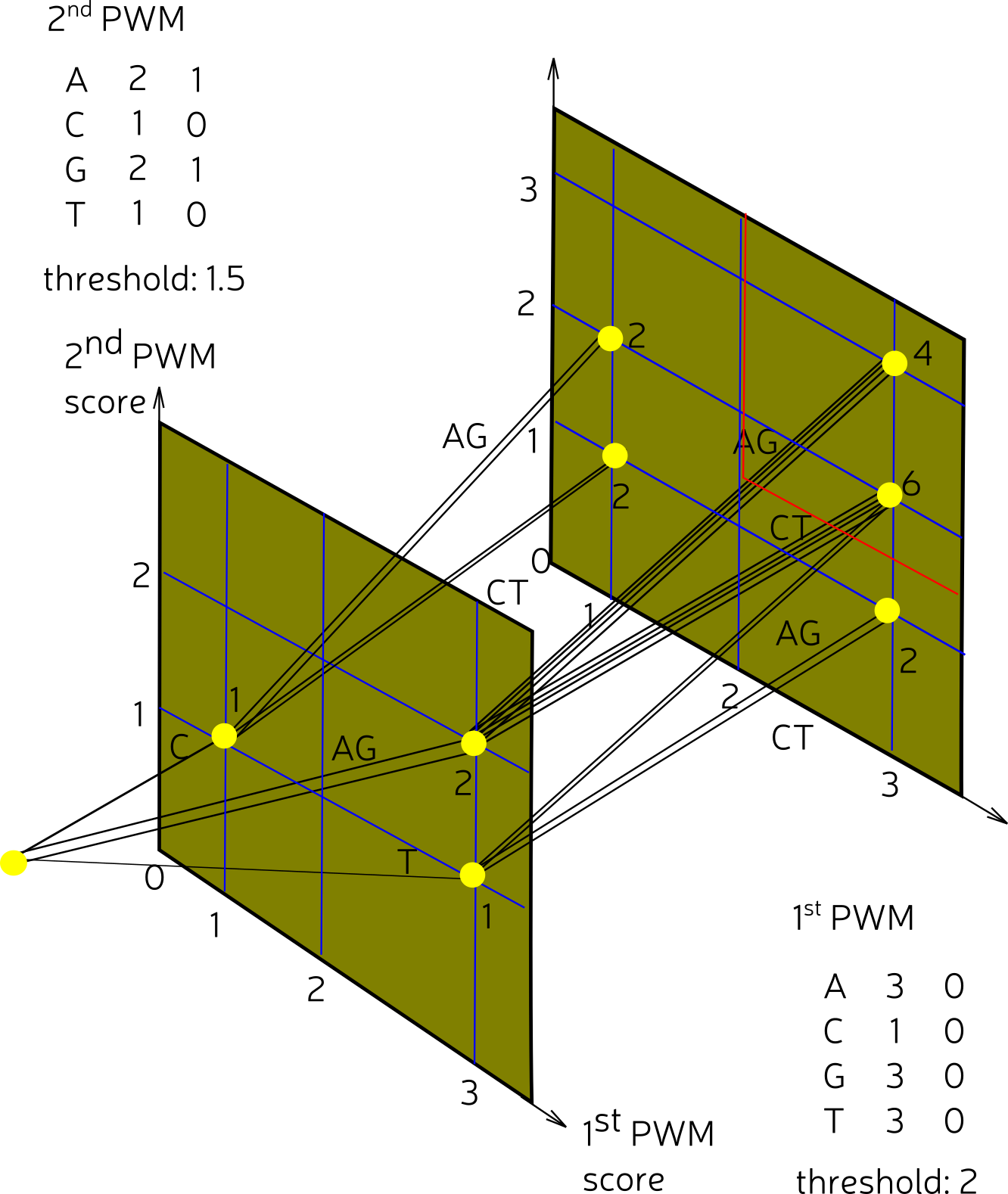
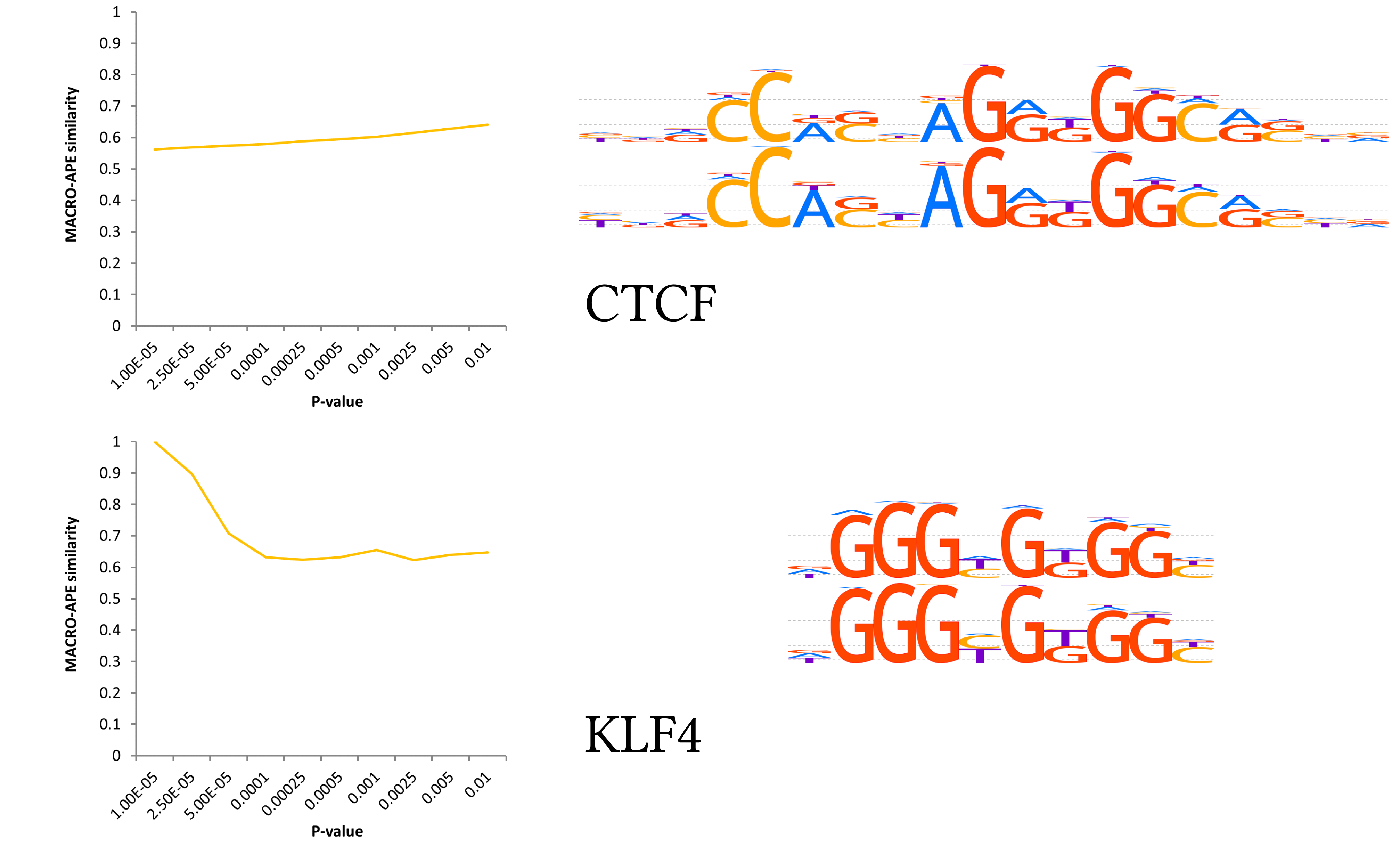 MACRO-APE: MAtrix CompaRisOn by Approximate P-value Estimation
MACRO-APE: MAtrix CompaRisOn by Approximate P-value Estimation
Насколько точно мотив описывает сайты связывания?
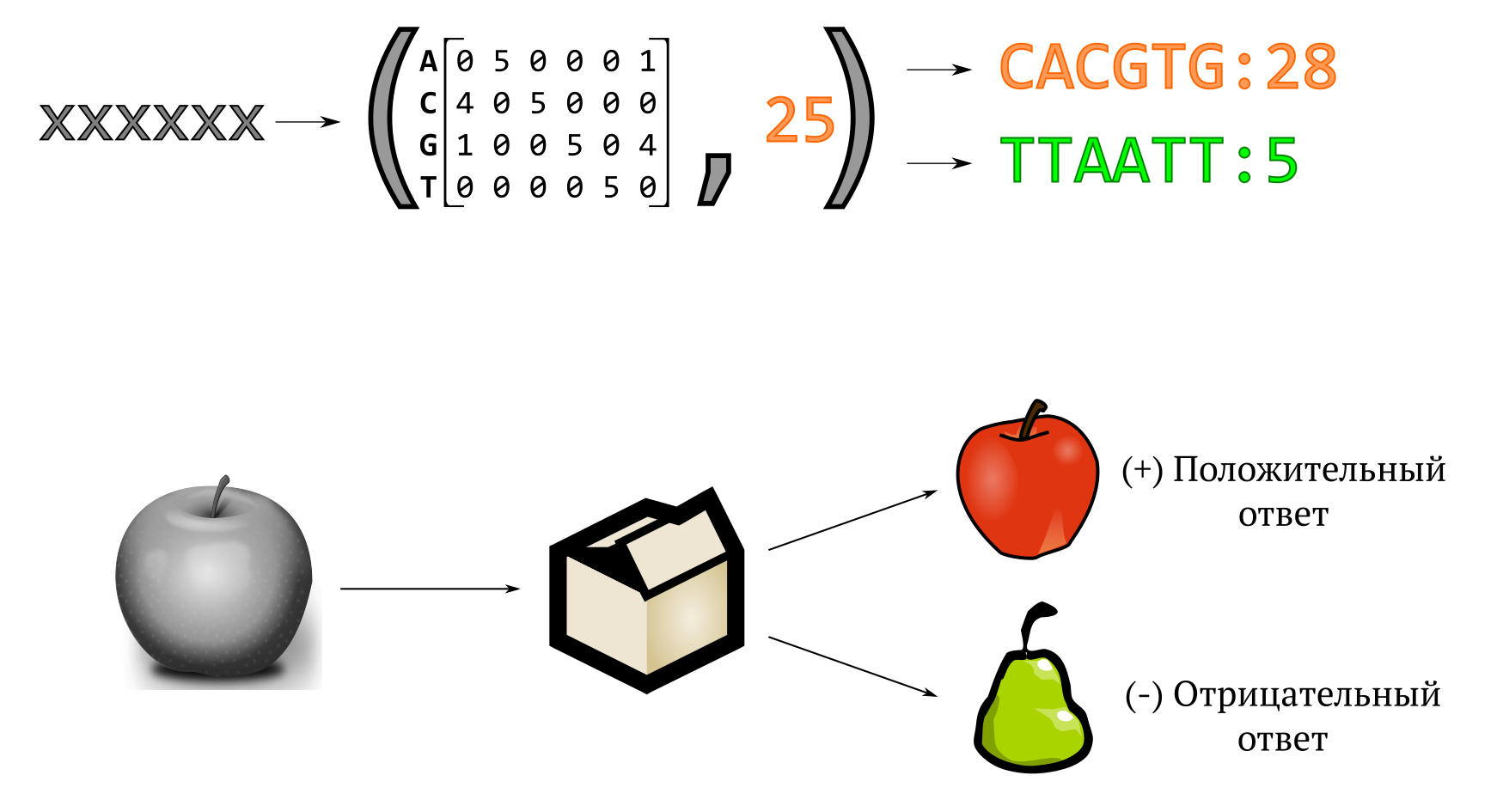
Мотив как бинарный классификатор
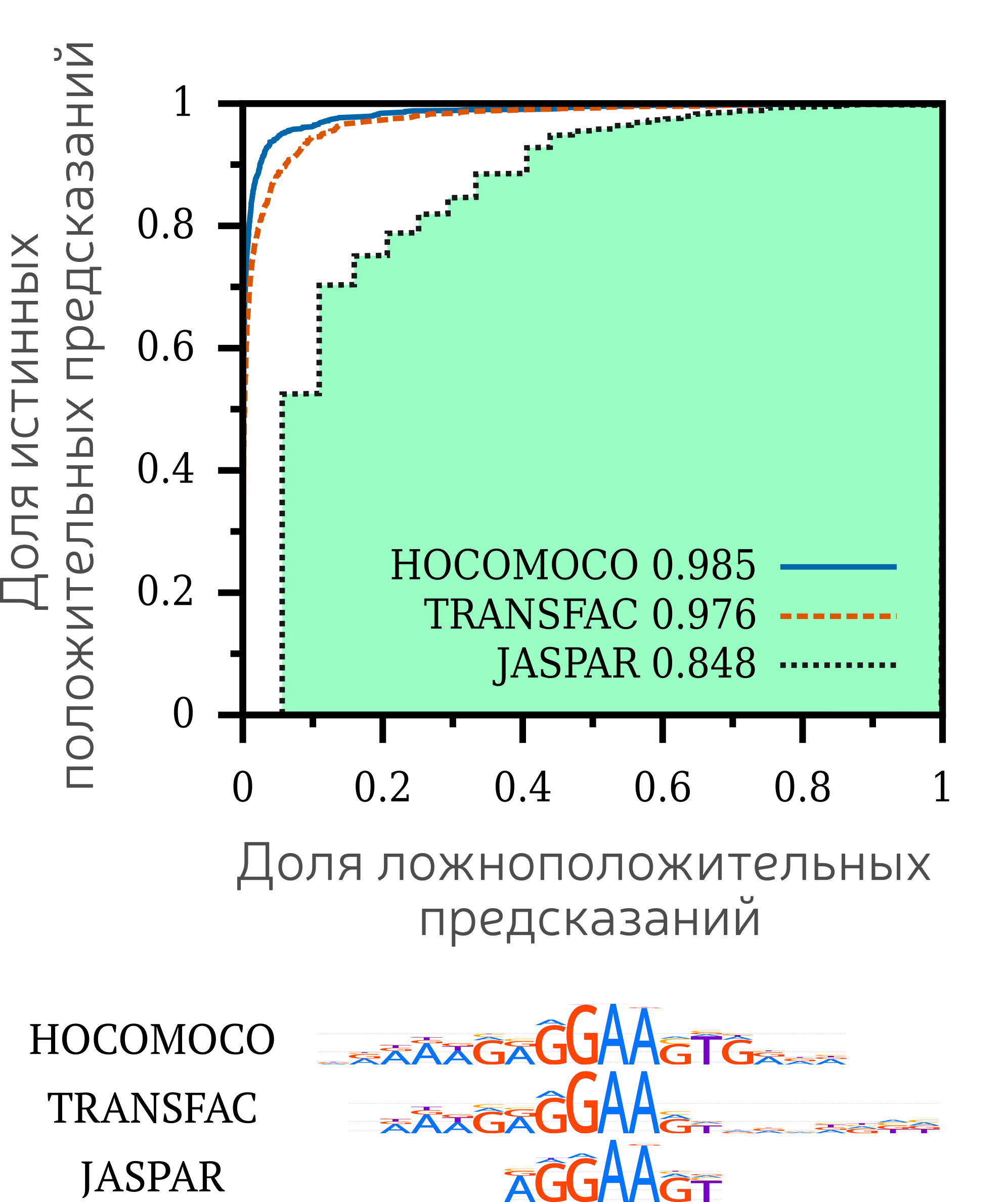
Сравнение качества моделей при помощи ROC-кривых
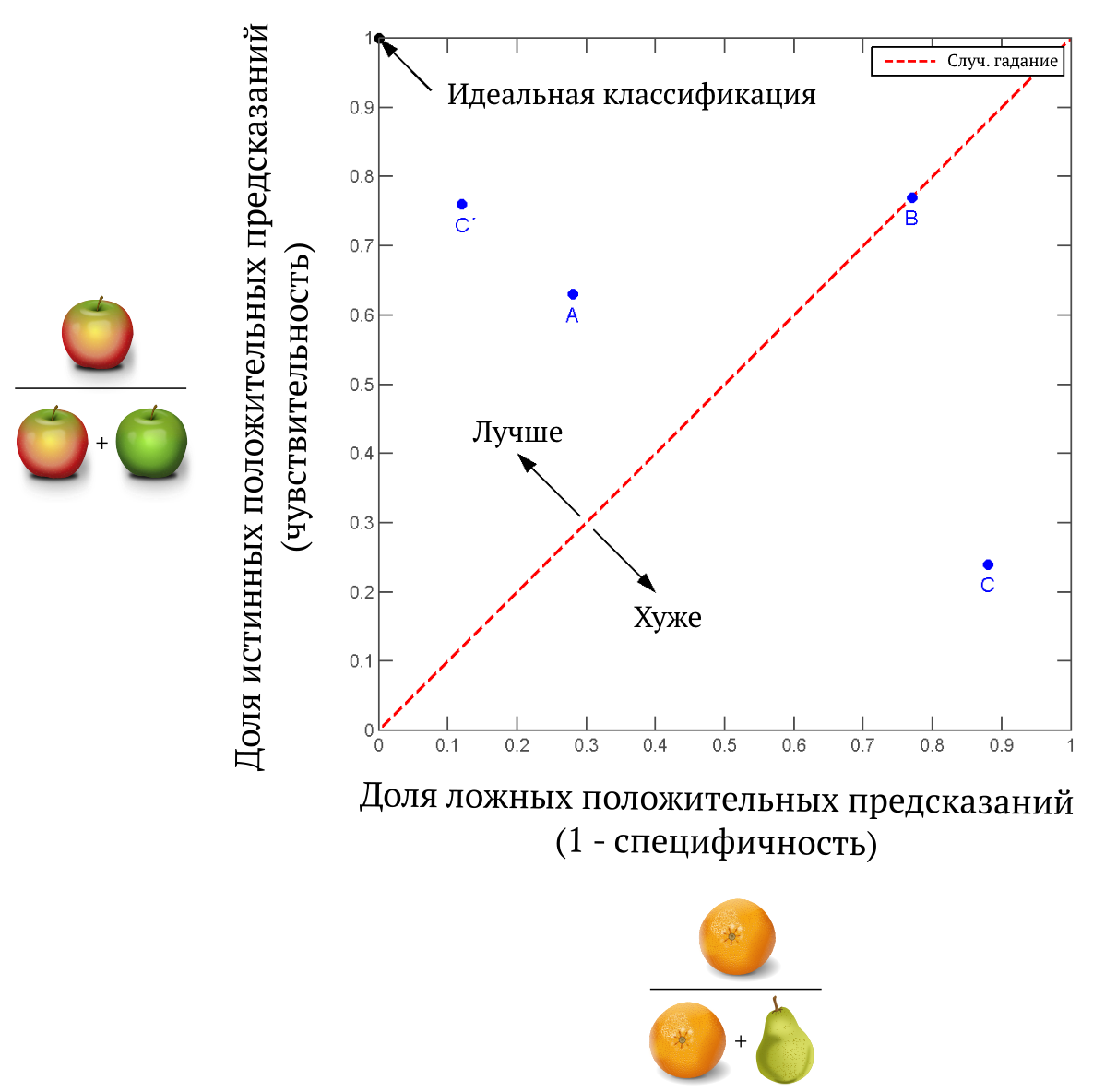
Сравнение качества моделей при помощи ROC-кривых
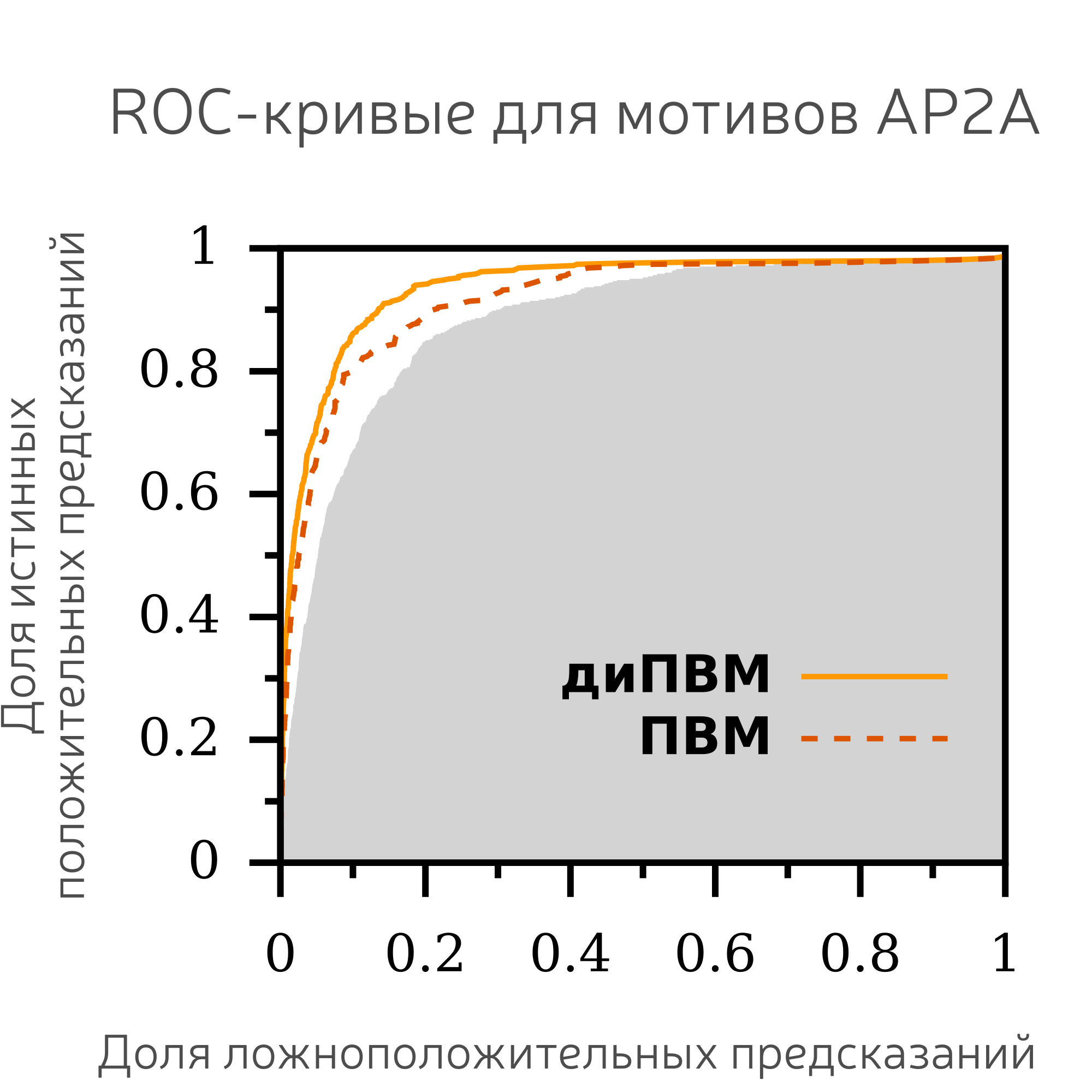
diChIPMunk - учитываем зависимости для соседних позиций

diChIPMunk - учитываем зависимости для соседних позиций

Развитие сложных моделей - учет зависимостей между удаленными позициями
- с помощью байесовских сетей или скрытых марковских моделей (BaMM, Slim, TFFM)
- с помощью решающих деревьев
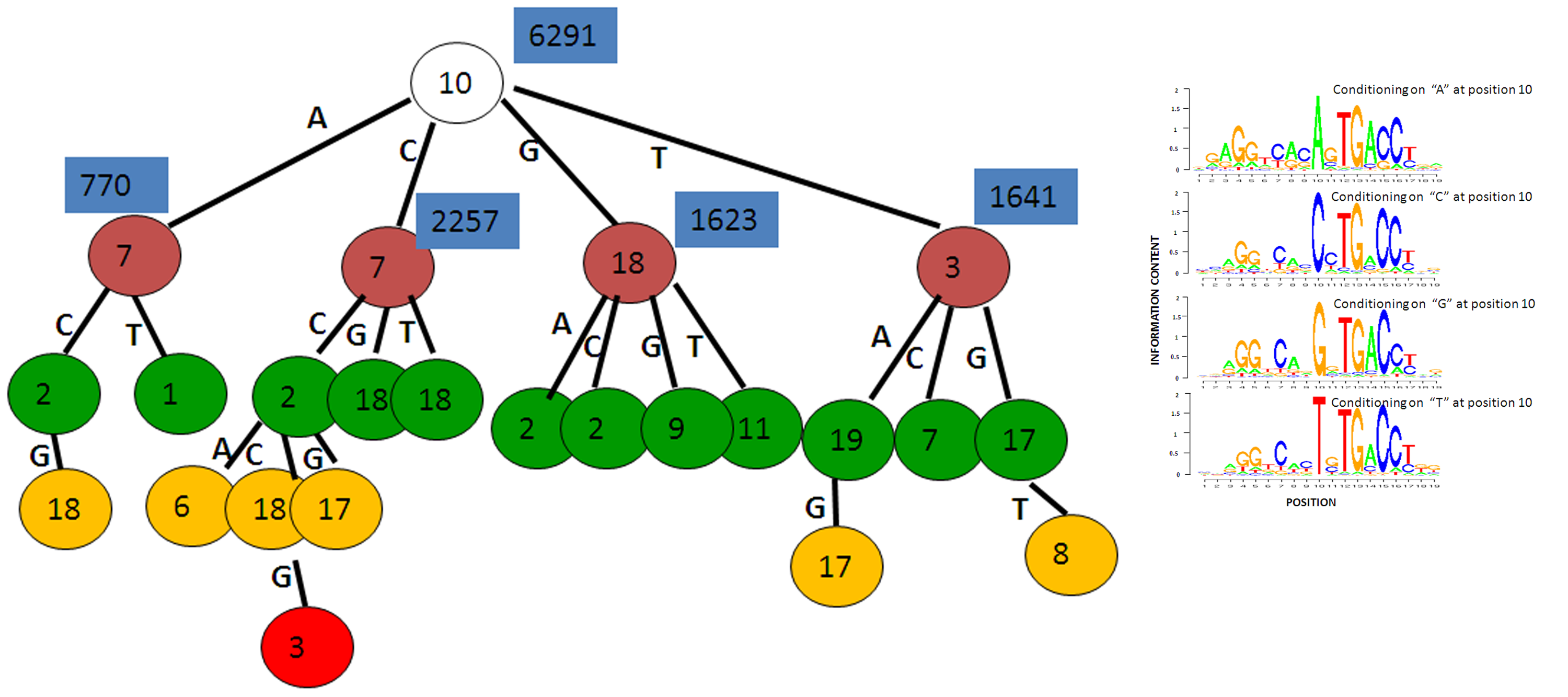 Bi et al., PLoS one, 2011
Bi et al., PLoS one, 2011
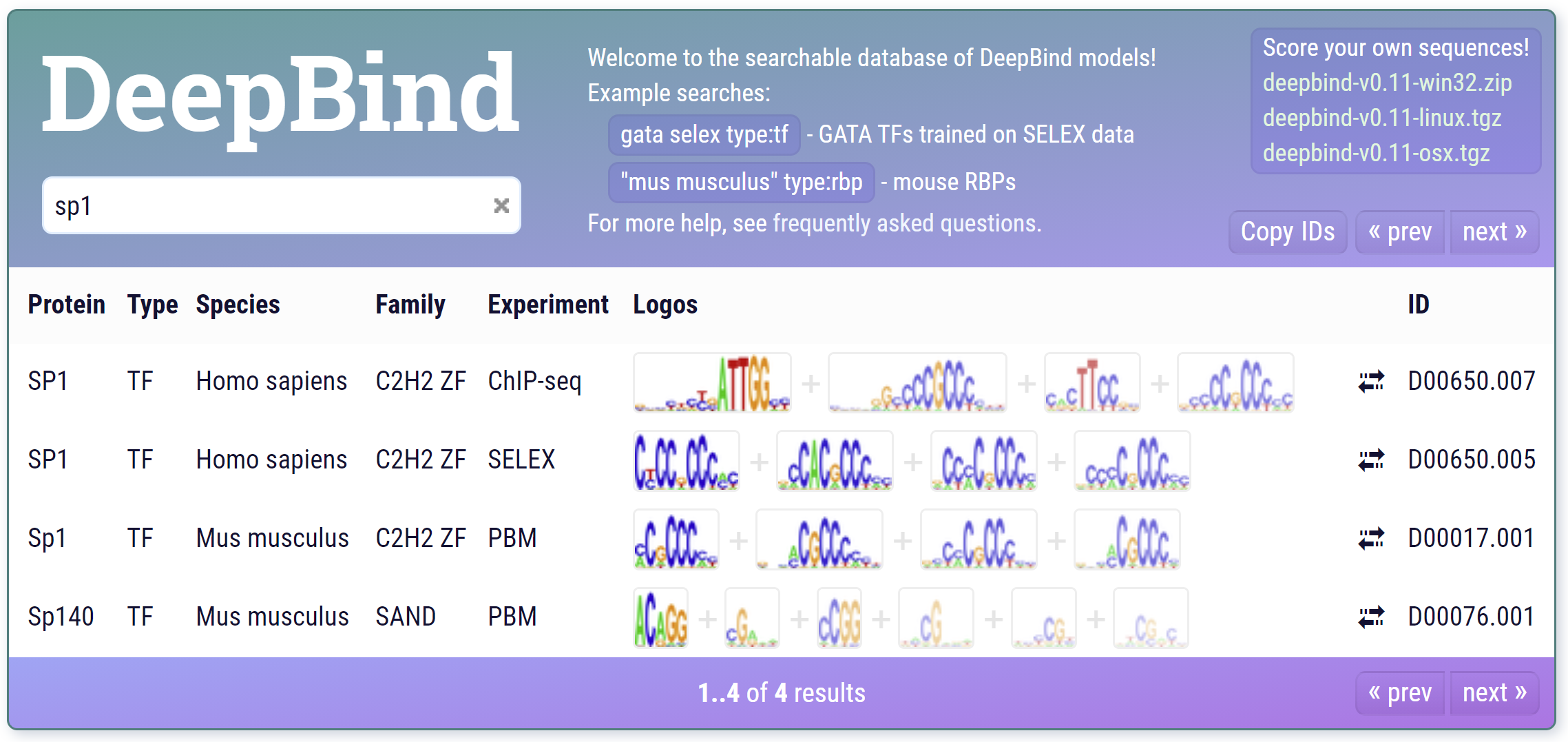
Искуственные нейронные сети!

DeepBind - первый успешный пример

Alipanahi et al. 2015

От мотивов к ключевым регуляторам
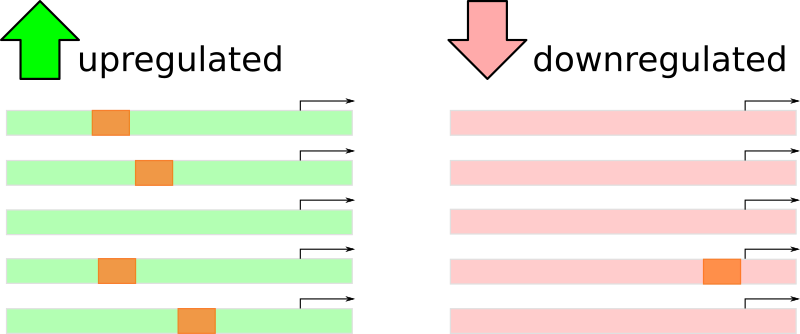

Не все так просто: грамматика определяет эффект
Ridinger-Saison et al., NAR, 2012
Не все так просто: грамматика определяет эффект

Boeva et al., Frontiers in Genetics, 2016
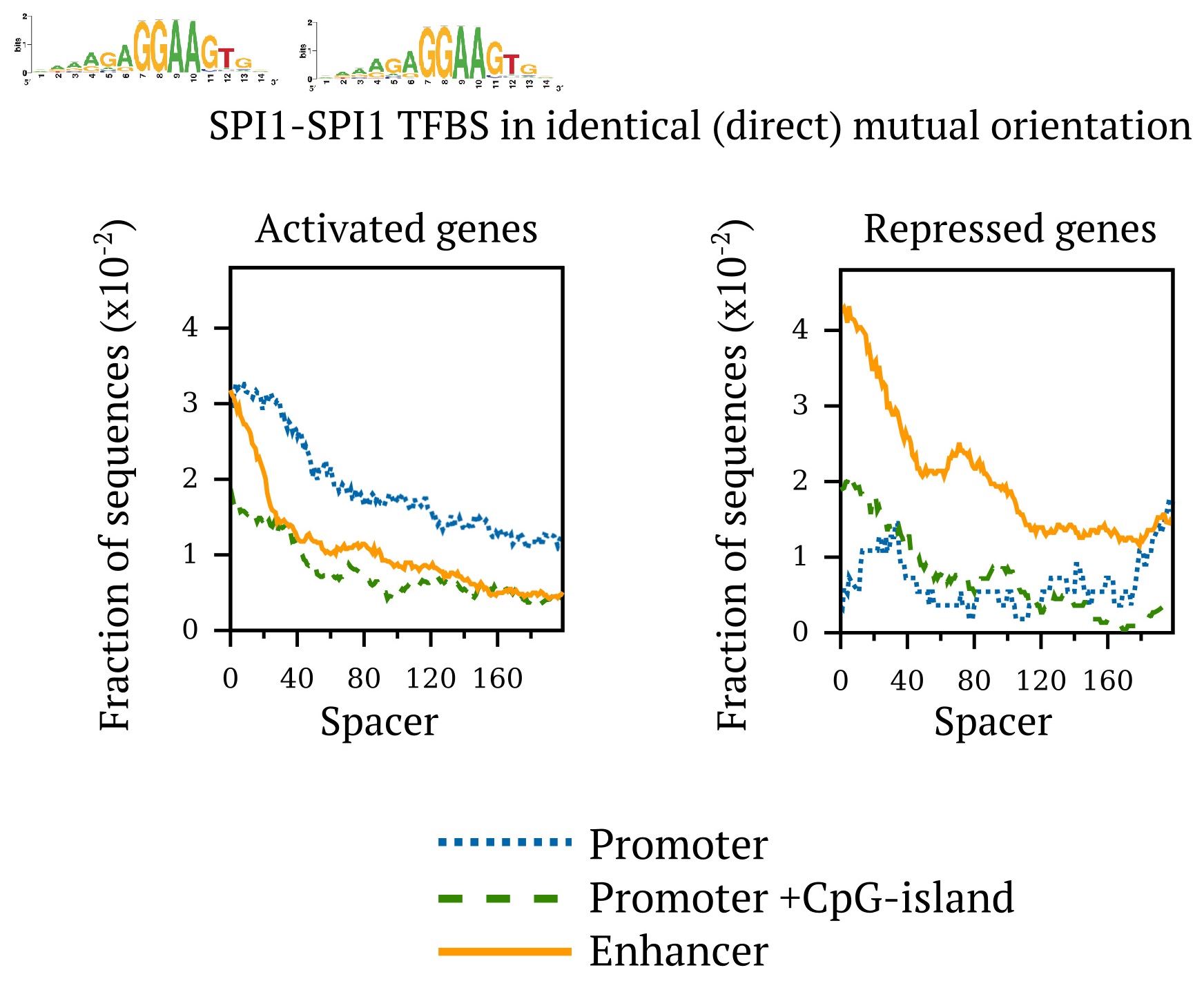
Мотив как инструмент для проверки адекватности ChIP-Seq
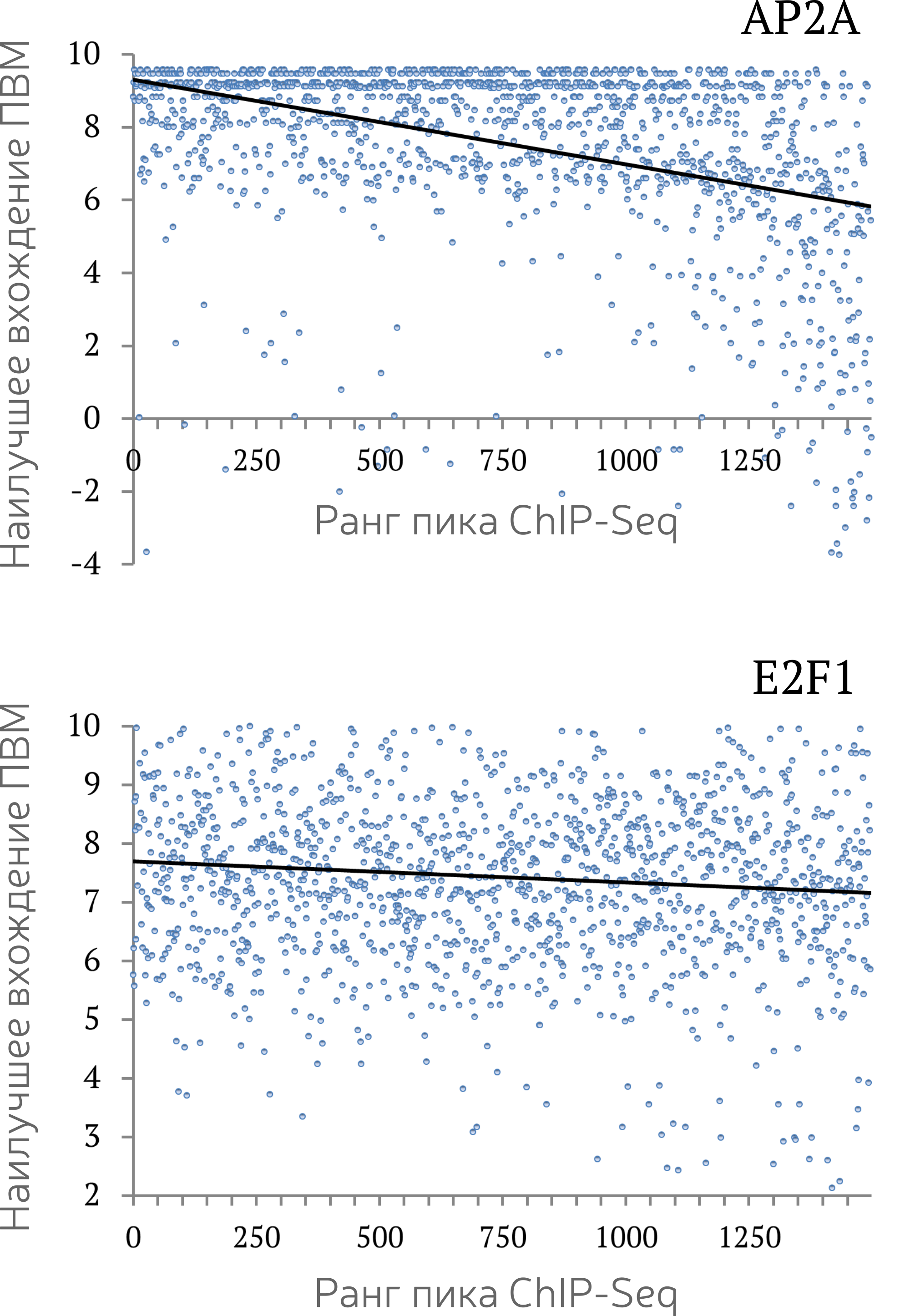
Kulakovskiy et al., 2013
Распределение вхождений мотивов коррелирует с профилем покрытия ChIP-Seq
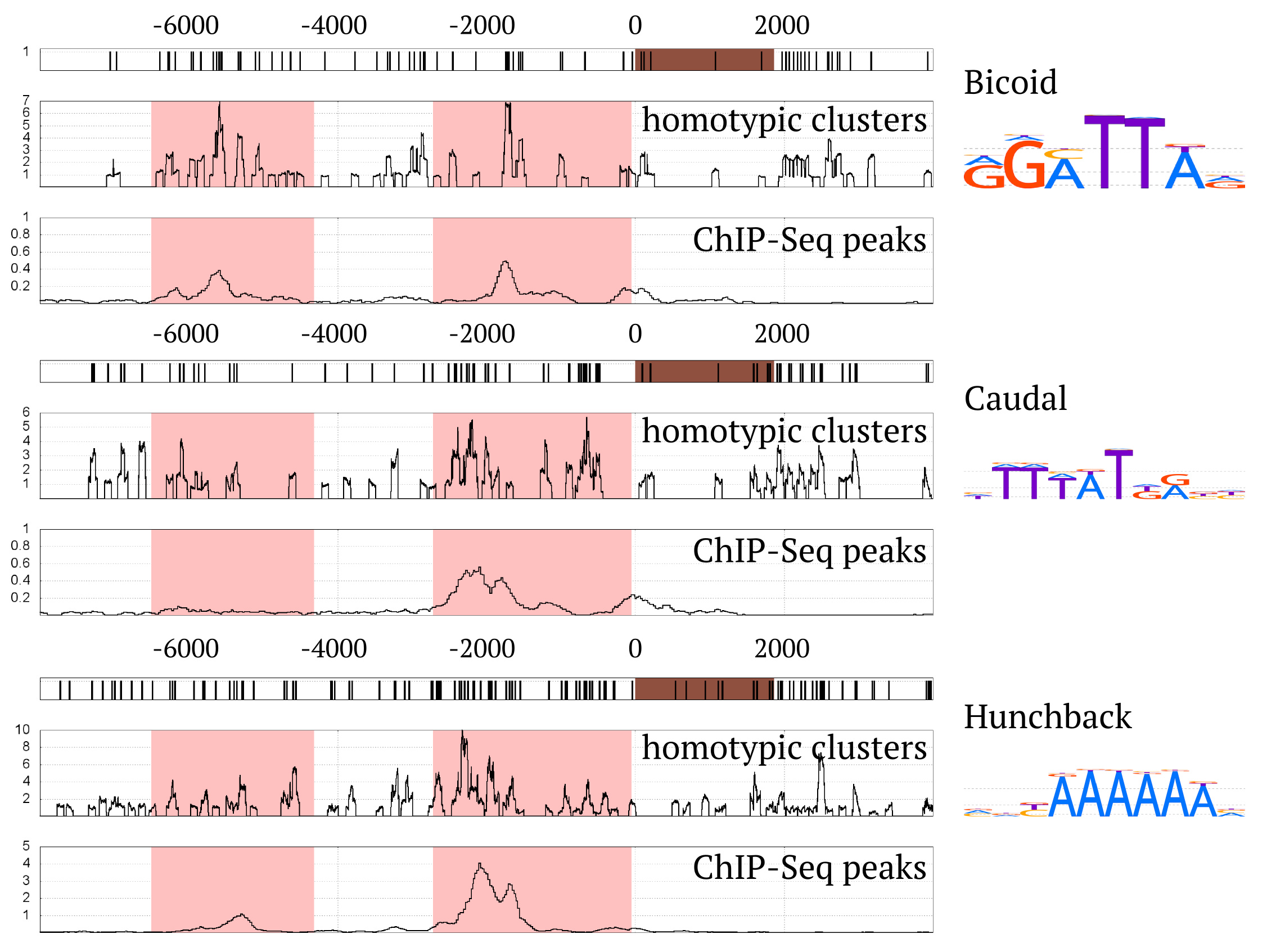
Kulakovskiy et al., 2013
Вхождения "правильного" мотива предпочитают вершины
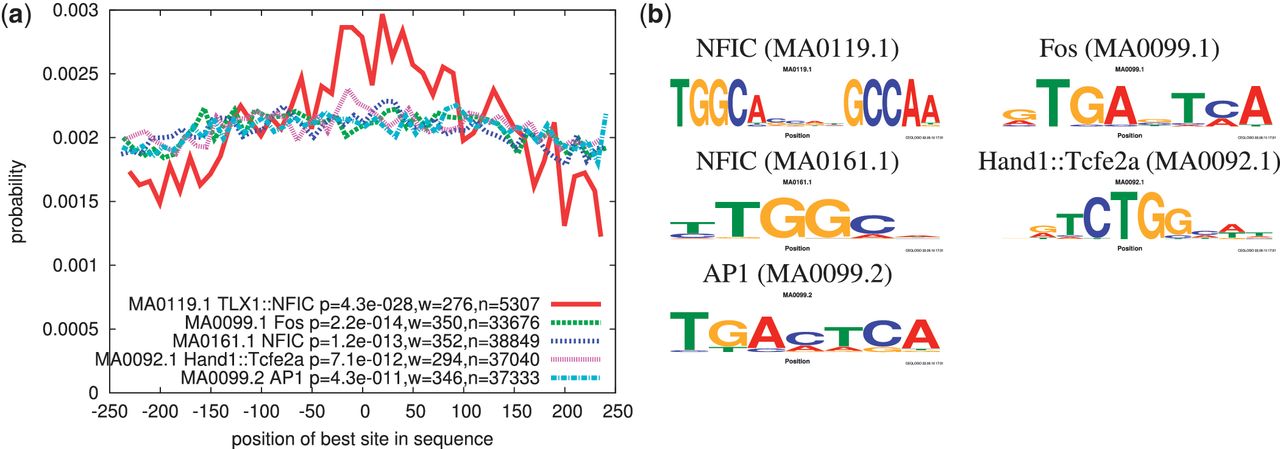
Bailey and Machanick, NAR, 2012
"Откуда мы"


ИОГен РАН - ИМБ РАН (Москва)
Лаборатория системной биологии и вычислительной генетики, Лаборатория ДНК-белковых взаимодействий

ИБ РАН (Пущино)
Группа регуляции биосинтеза белка
И кто мы

Shameless self-promotion: autosome.ru software
| Motif discovery with ChIPMunk (for CHromatin ImmunoPrecipitation) |  |
| Computing Jaccard Similarity of motifs with MACRO-APE (MAtrix CompaRisOn by Approximate P-value Estimation) |  |
| Fast motif finding with SPRY-SARUS (Straightforward yet Powerful Rapid SuperAlphabet Representation Utilized for motif Search) |  |
| Interactive motif finding with MoLoTool (Transcription Factor Motif Location Toolbox) |  |
| Functional annotation of regulatory variants with PERFECTOS-APE (PrEdicting Regulatory Functional Effect by Approximate P-value Estimation) |  |
Что мы знаем про мотив-ом человека?
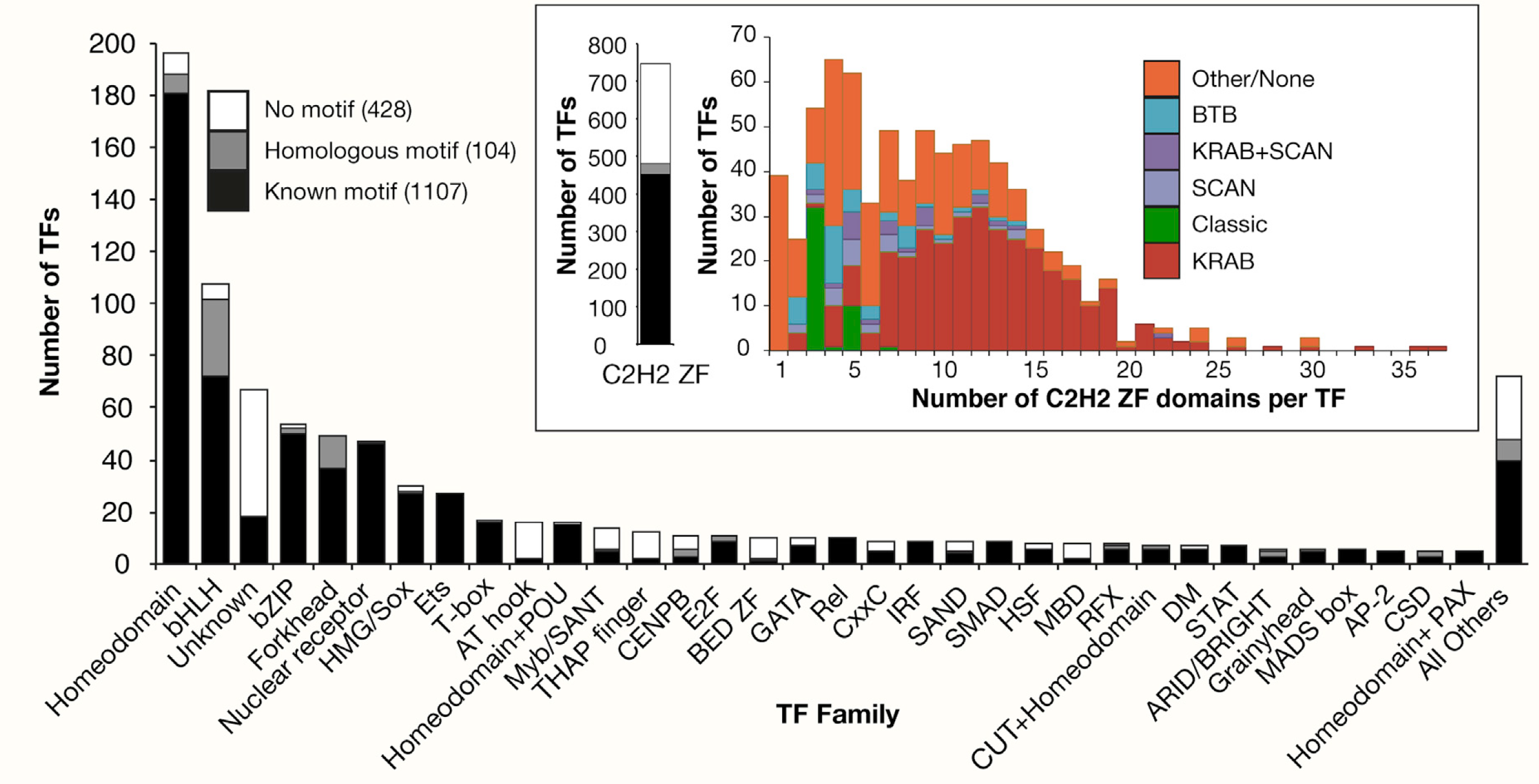 Оптимистичная оценка из Lambert et al., 2018
Оптимистичная оценка из Lambert et al., 2018
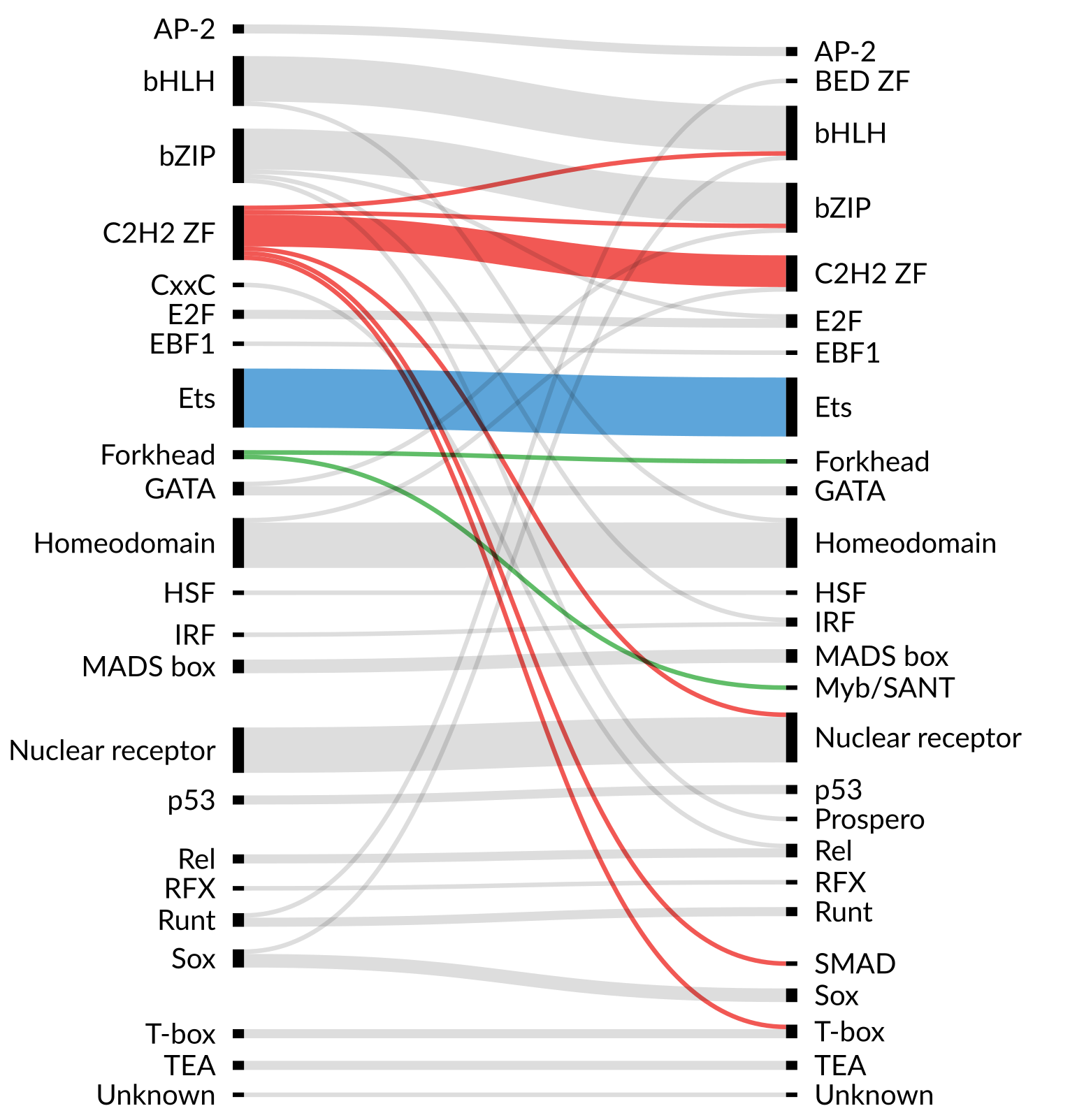
Ambrosini et al., 2020, Genome Biol
Мотив-ом практически полон
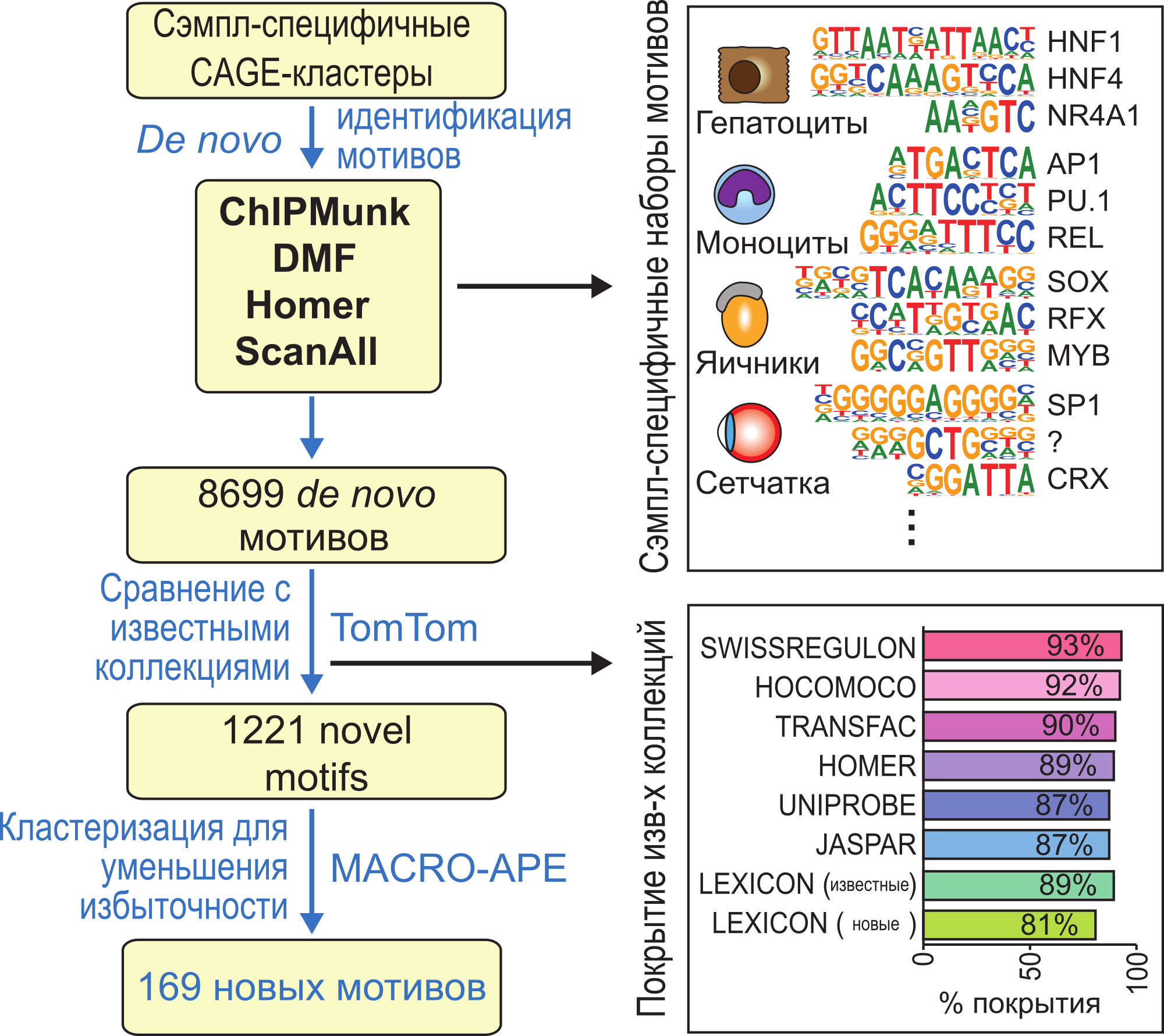
Take-home

Анализ мотивов - любимая задача биоинформатиков
Глубокое обучение уже тут, остается его аккуратно применить
Весовые матрицы как модели специфичности остаются полезными